시계열 분석이란?
시간에 따라 측정된 데이터의 패턴, 동향 및 예측을 이해하기 위한 통계적인 분석 방법으로 시계열 데이터로부터 추세, 계절성, 주기성 등의 패턴을 식별하고, 데이터의 특성을 이해하며, 예측 모델을 구축할 수 있다.
시계열 분석에는 다양한 기법들이 사용되며, 이 중 일반적으로 사용되는 기법으로는 시계열 그래프 시각화, 시계열 분해, 이동평균, 지수평활법, ARIMA 모델, 지수평활 이동평균법(ETS), 회귀분석, 신경망 등이 있습니다. 이러한 기법들을 사용하여 시계열 데이터를 분석하고 예측함으로써 경제, 금융, 기상, 주식 등 다양한 분야에서 유용하게 활용됩니다.
1. library
library(readr)
library(dplyr)
library(tidyr)
library(stringr)
library(tidyverse)2. 데이터 전처리
- 사용 데이터: CSSE Covid 19
- 대상 기간: 2020.03 ~ 2022.07 (일별 데이터)
⭐ 전처리 과정에 대한 자세한 설명은 탐색적 데이터 분석의 3. 일별 데이터 기준 참고 부탁드립니다.
1) 로드 및 로우데이터 생성
# (1) 데이터 불러오기
getwd()
setwd("C:/Rwork/csse_covid_19_daily_reports")
korea20 <- list.files (pattern = '*2020.csv') %>% .[c(40:345)]
korea21 <- list.files (pattern = '*2021.csv')
korea22 <- list.files (pattern = '*2022.csv') %>% .[c(1:212)]
# (2) 2020-03-01 daily 값 추출 & 구하고자 하는 기준일과의 비교용 변수
compKo_origin <- read.csv('02-29-2020.csv')
compKo_pre <- compKo_origin %>% filter(Country.Region=='South Korea') %>% select(Country.Region, Confirmed, Deaths) %>% rename("Country_Region" = "Country.Region")
compKo_pre[1,1] <- 'Korea, South'
# (3) for문을 통해 얻은 일일값 담을 변수 생성
dailyKo1 <- compKo_pre[ ,-c(2,3)] %>% as.data.frame() %>% rename("Country_Region" = ".")
dailyKo2 <- compKo_pre[ ,-c(2,3)] %>% as.data.frame() %>% rename("Country_Region" = ".")2) 반복문으로 일별 데이터 추출
본 과정에서 사용한 파일(데이터셋)의 2021년 중 일부는 컬럼명(CountryRegion) 및 국가명(Korea, South)이 상이하므로 기존과는 아주 조금 차이가 있습니다. 추후 데이터 프레임 병합을 하기 위해 컬럼명 및 국가명을 통일 해주어야 하므로 if문을 활용하여 추출하는 과정에서 변경해주어 추후 다시 변경해야 하는 번거로움이 없게 해줍니다.
그 외의 부분은 앞서 말했던 탐색적 데이터 분석과 동일하므로 설명은 생략하도록 하겠습니다.
# (4) '기준일의 일일수치 == (기준일 - 전일)' 확인을 위한 반복문
# - 2020 (03 ~ 12)
for(i in 1:length(korea20)){
comKo1 <- read.csv(korea20[i])
if(i <= 21) {
if(i < 10){
comKo2 <- comKo1 %>% filter(Country.Region=='South Korea') %>% select(Country.Region, Confirmed, Deaths) %>% rename("Country_Region" = "Country.Region")
comKo2[1,1] <- 'Korea, South'
} else if(i == 10) {
comKo2 <- comKo1 %>% filter(Country.Region=='Republic of Korea') %>% select(Country.Region, Confirmed, Deaths) %>% rename("Country_Region" = "Country.Region")
comKo2[1,1] <- 'Korea, South'
} else {
comKo2 <- comKo1 %>% filter(Country.Region=='Korea, South') %>% select(Country.Region, Confirmed, Deaths) %>% rename("Country_Region" = "Country.Region")
}
} else {
comKo2 <- comKo1 %>% filter(Country_Region=='Korea, South') %>% select(Country_Region, Confirmed, Deaths)
}
dump <- merge(comKo2, compKo_pre, by="Country_Region", all=T)
dump$daily <- dump$Confirmed.x - dump$Confirmed.y
dump$daily2 <- dump$Deaths.x - dump$Deaths.y
dailyKo1 <- merge(dailyKo1, dump[c(1,6)], by='Country_Region', all=T)
dailyKo2 <- merge(dailyKo2, dump[c(1,7)], by='Country_Region', all=T)
compKo_pre <- comKo2
}
# - 2021 및 2022의 반복문은 앞서 안내 드린 '탐색적 데이터 분석' 과정과 동일하므로 생략합니다.3) 최종 일별 데이터
이번 단계에서는 기존과 조금 다른 부분이 있어 설명 드립니다.
날짜 추출은 파일명에서 하지 않고 as.Date() 와 seq() 함수를 사용하여 쉽고 간단하게 생성하였습니다. 또한, 추후 다양한 목적에 사용하기 위해 '확진자와 사망자의 값을 합친 데이터프레임', '확진자 데이터 프레임', '사망자 데이터 프레임' 총 3개를 생성합니다.
# (5) 값만 추출(나라이름 제외) %>% 데이터프레임형식으로 전환
con <- dailyKo1 %>% .[ ,c(2:884)] %>% as.data.frame()
dea <- dailyKo2 %>% .[ ,c(2:884)] %>% as.data.frame()
# (6) 구하고자 하는 기간의 날짜
seqDate <- seq(as.Date("2020-03-01"), as.Date("2022-07-31"), by="day") %>% as.data.frame()
# (7-1) (5)과 (6) 열병합 후, 열 이름 변경 & 행 이름 제거
finalKo <- cbind(seqDate, t(con), t(dea))
colnames(finalKo) <- c("Day", "Confirmed", "Deaths")
rownames(finalKo) <- NULL
# >> 데이터 및 데이터 내 형식 확인
View(finalKo); str(finalKo)
head(finalKo)
# (7-2) 확진자와 사망자 분류하여 열병합 후, 열 이름 변경 & 행 이름 제거
finalCon <- cbind(seqDate, t(con))
finalDea <- cbind(seqDate, t(dea))
colnames(finalCon) <- c("Day", "Confirmed")
rownames(finalCon) <- NULL
colnames(finalDea) <- c("Day", "Deaths")
rownames(finalDea) <- NULL
# >> 데이터 및 데이터 내 형식 확인
View(finalCon); str(finalCon)
View(finalDea); str(finalDea)▼ Console of head(finalKo) ▼
| Korea_dailyCon | Korea_dailyDea | |
|---|---|---|
| 2020-03-01 | 586 | 1 |
| 2020-03-02 | 599 | 11 |
| 2020-03-03 | 851 | 0 |
| 2020-03-04 | 435 | 7 |
| 2020-03-05 | 467 | 0 |
▼ Console of head(finalCon) ▼
| Day | Confirmed | |
|---|---|---|
| 1 | 2020-03-01 | 586 |
| 2 | 2020-03-02 | 599 |
| 3 | 2020-03-03 | 851 |
| 4 | 2020-03-04 | 435 |
| 5 | 2020-03-05 | 467 |
▼ Console of head(finalDea) ▼
| Day | Deaths | |
|---|---|---|
| 1 | 2020-03-01 | 1 |
| 2 | 2020-03-02 | 11 |
| 3 | 2020-03-03 | 0 |
| 4 | 2020-03-04 | 7 |
| 5 | 2020-03-05 | 0 |
3. 추세선 확인
1) library
깔끔하고 다양한 효과를 줄 수 있는 ggplot2와 plots 패널에 다수의 ggplot 그래프를 한번에 출력해주는 gridExtra를 library 합니다.
library(ggplot2)
library(gridExtra)2) 대상기간 내 확진자 및 사망자 추세
# 확진자
entCon <- ggplot(data = finalKo, aes(x=Day, y=Confirmed)) + geom_line(color="#0055FF", size = 1) +
geom_smooth(color="yellow", size = 0.5) +
labs(title = "Covid-19 Confirmed in Korea", subtitle = "from 03-2020 to 07-2022")
# 사망자
entDea <- ggplot(data = finalKo, aes(x=Day, y=Deaths)) + geom_line(color="red", size = 1) +
geom_smooth(color="yellow", size = 0.5) +
labs(title = "Covid-19 Death in Korea", subtitle = "from 03-2020 to 07-2022")
# 2003 ~ 2207 기간 그래프
grid.arrange(entCon, entDea, nrow=2, ncol=1) ▼ Console ▼
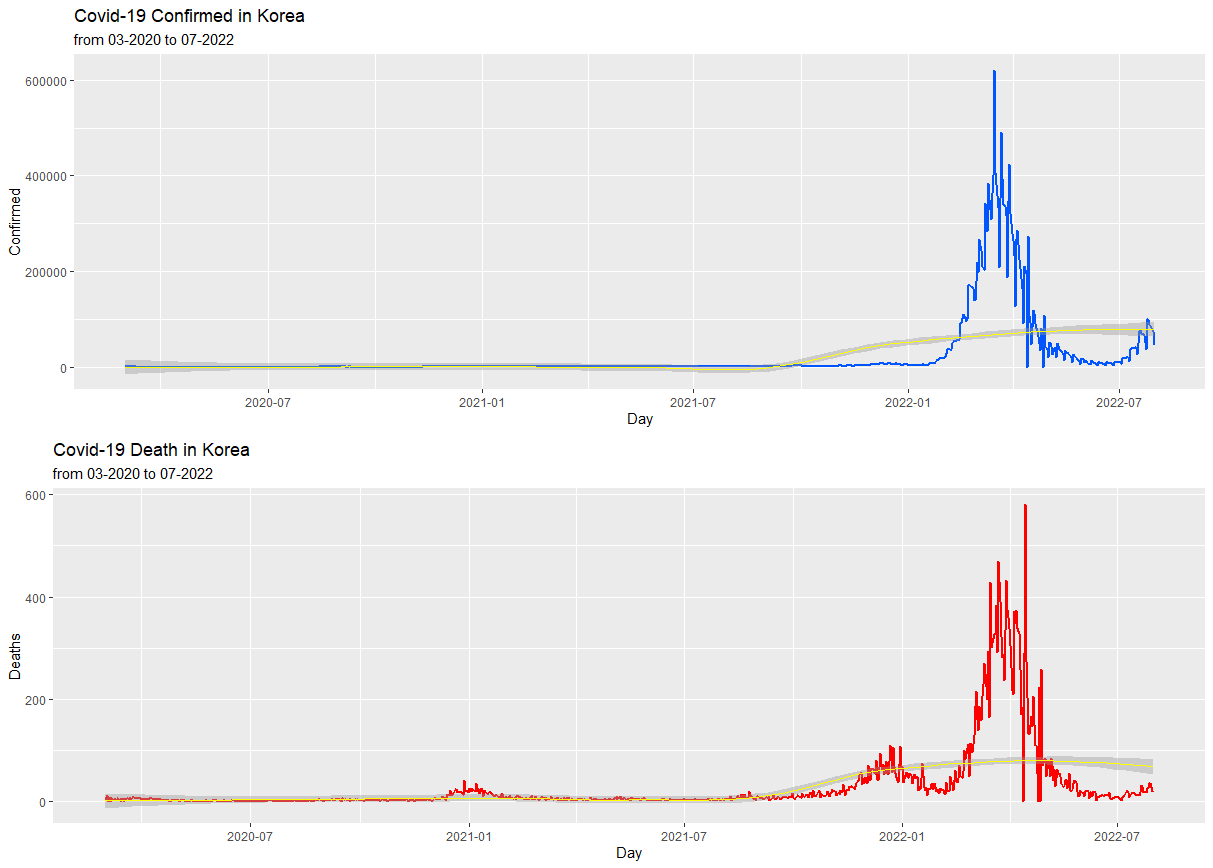
> 2021년 약 3월부터 5월까지 가파른 추세로 상승했다가 하락하는 것을 알 수 있습니다. 해당 구간의 값이 다른 구간에 비해 지나치게 큰 값들이 분포해 있으므로 연도별로 구분하여 시각화 그래프를 만들어 전체적인 추세를 확인하겠습니다.
3) 연도별 확진자 및 사망자 추세
# 20년도
part20_Con <- ggplot(data = finalKo[c(1:306), ], aes(x=Day, y=Confirmed)) + geom_line(color="#0055FF", size = 1) +
geom_smooth(color="yellow", size = 0.5) +
labs(title = "Covid-19 Confirmed in Korea", subtitle = "from 03-2020 to 12-2020")
part20_Dea <- ggplot(data = finalKo[c(1:306), ], aes(x=Day, y=Deaths)) + geom_line(color="red", size = 1) +
geom_smooth(color="yellow", size = 0.5) +
labs(title = "Covid-19 Death in Korea", subtitle = "from 03-2020 to 12-2020")
# 21년도
part21_Con <- ggplot(data = finalKo[c(307:671), ], aes(x=Day, y=Confirmed)) + geom_line(color="#0055FF", size = 1) +
geom_smooth(color="yellow", size = 0.5) +
labs(title = "Covid-19 Confirmed in Korea", subtitle = "from 01-2021 to 12-2021")
part21_Dea <- ggplot(data = finalKo[c(307:671), ], aes(x=Day, y=Deaths)) + geom_line(color="red", size = 1) +
geom_smooth(color="yellow", size = 0.5) +
labs(title = "Covid-19 Death in Korea", subtitle = "from 01-2021 to 12-2021")
# 22년도
part22_Con <- ggplot(data = finalKo[c(672:883), ], aes(x=Day, y=Confirmed)) + geom_line(color="#0055FF", size = 1) +
geom_smooth(color="yellow", size = 0.5) +
labs(title = "Covid-19 Confirmed in Korea", subtitle = "from 01-2022 to 07-2022")
part22_Dea <- ggplot(data = finalKo[c(672:883), ], aes(x=Day, y=Deaths)) + geom_line(color="red", size = 1) +
geom_smooth(color="yellow", size = 0.5) +
labs(title = "Covid-19 Death in Korea", subtitle = "from 01-2022 to 07-2022")
# 연도별 그래프
grid.arrange(part20_Con, part20_Dea, part21_Con, part21_Dea, part22_Con, part22_Dea, nrow=3, ncol=2) ▼ Console ▼
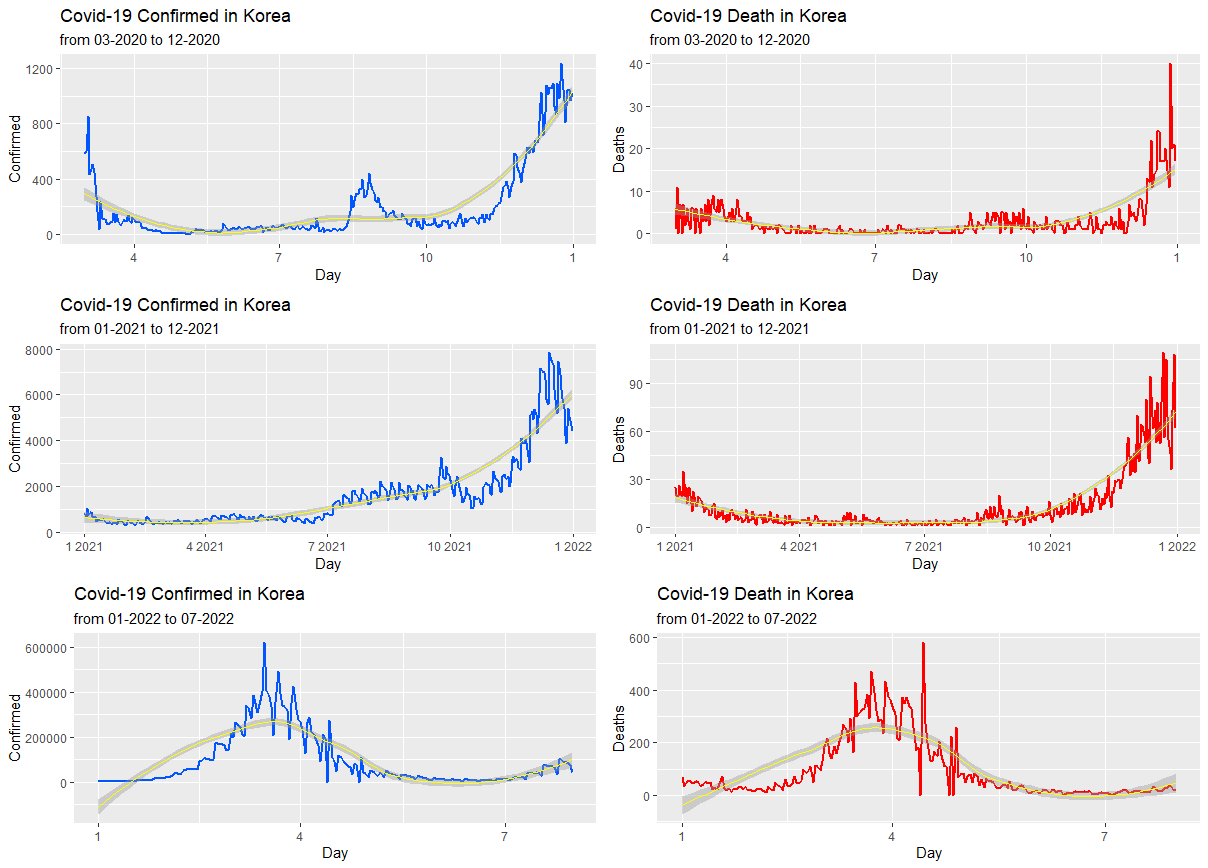
> 대상 기간 내 확진자 및 사망자의 추세를 연도별로 구분하였습니다. 전체 기간의 그래프에 비해 추세를 파악하기 한결 쉬워졌습니다.
그래프를 보았을 때, 확진자 및 사망자 모두 시간이 경과할 수록 계속해서 증가해가다가 2022년 약 2~3월 부터 수가 가파르게 증가하기 시작 하더니 확진자는 약 3~4월 사이, 사망자는 약 3월 말~5월 사이에 급증하며 최고치를 기록합니다.
y축 수의 차이를 확인하면 상승 폭이 다른 연도에 비해 굉장히 큰 것을 알 수 있습니다. 이 그래프의 데이터가 코로나임을 감안하면 가파른 상승폭에 원인을 21년 말 발생하여 22년 초 대유행한 오미크론으로 짐작할 수 있습니다.
4. 4가지 변동요인 분해
1) library
시계열 분석을 위해서 데이터의 type을 변환해주어야 하므로 아래의 package를 library 합니다.
library(zoo) # 데이터프레임 > 시계열 형식으로 변환
library(lubridate) # 날짜와 시간 데이터를 처리
library(forecast) # 시계열 데이터의 예측과 관련 기능을 제공2) 데이터 type 변환
library(zoo)
zooCon <- read.zoo(finalCon)
zooDea <- read.zoo(finalDea)
# 날짜의 주기를 1년 단위로 설정하여 처리
library(lubridate)
tsCon <- as.ts(zooCon) %>% ts(start = decimal_date(as.Date("2020-03-01")), frequency = 365)
tsDea <- as.ts(zooDea) %>% ts(start = decimal_date(as.Date("2020-03-01")), frequency = 365)3) 시각화
library(forecast)
options(scipen=100)
tsCon %>% stl(t.window = 13, s.window = "periodic", robust=T) %>% autoplot(main="Time Sereies of Confirmed")
tsDea %>% stl(t.window = 13, s.window = "periodic", robust=T) %>% autoplot(main="Time Sereies of Deaths")(1) 확진자
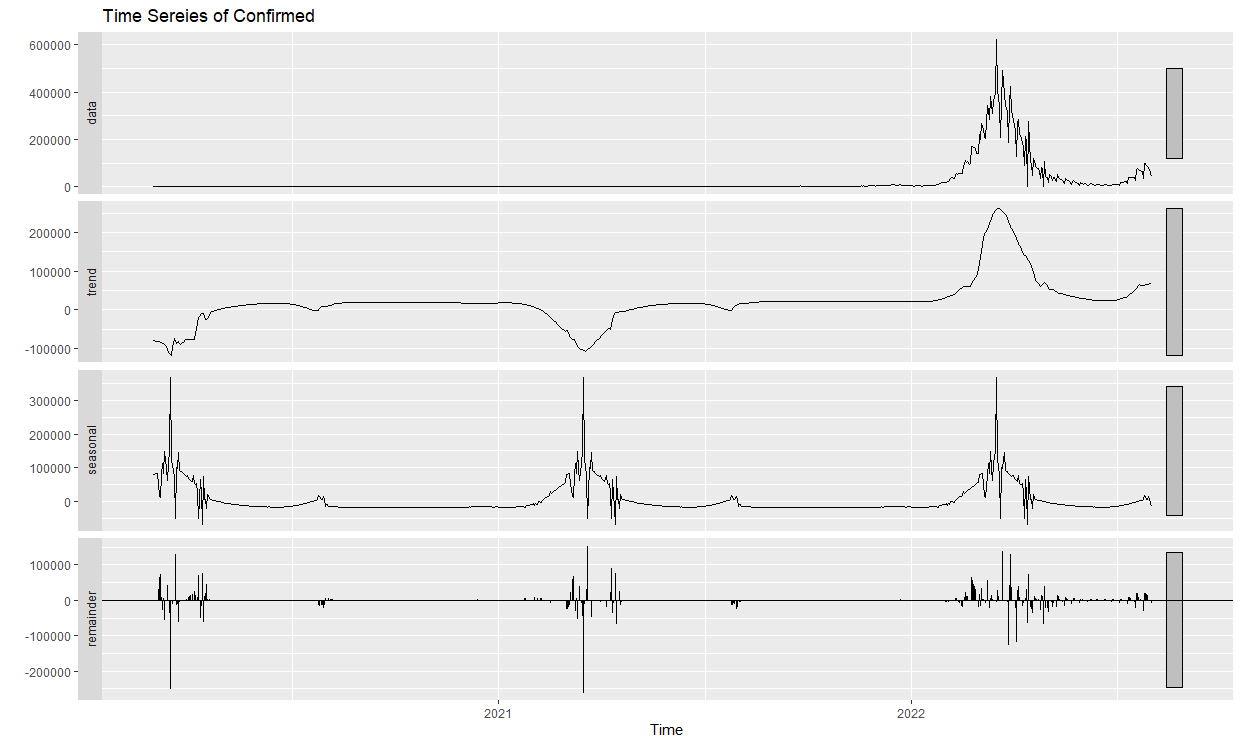
(2) 사망자
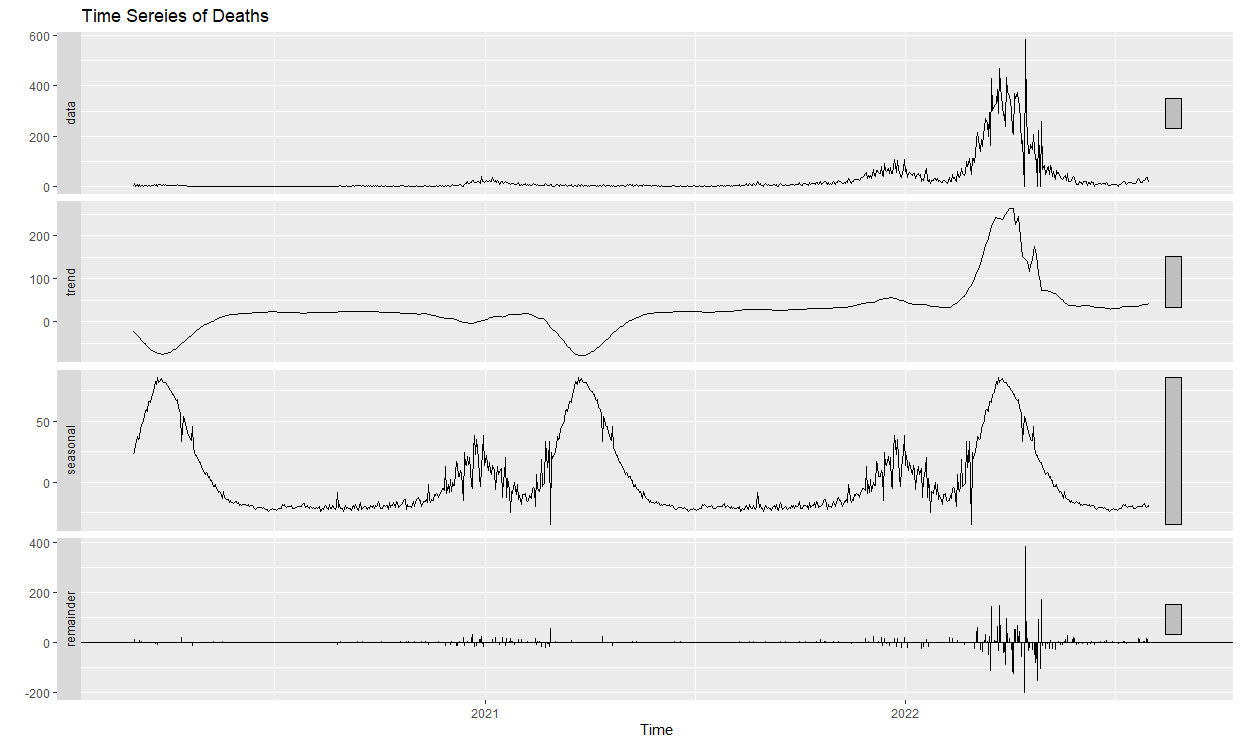
4) 해석
trend(추세)를 보았을 때 확진자 및 사망자 가끔 감소하는 경향이 있지만 대체로 증가하는 추세를 보이며 22년 봄 계절에는 오미크론의 영향으로 급증한 후 해소된 것을 알 수 있습니다. 이를 통해 대한민국 확진자 및 사망자는 앞으로도 계속 발생할 것을 예측할 수 있습니다.
seasonal(계절성)을 보았을 때 특정 주기에 반복적인 패턴을 보입니다. 이는 계절성을 갖고 있는 것으로 여름 보다는 선선한 계절에 보다 발생률이 높은 것을 알 수 있습니다. 이를 통해 코로나 바이러스는 덥고 습한 때보다 선선하고 추운 계절에 더 전파가 잘 된다는 것을 예측할 수 있습니다.
Remainder(나머지 성분)을 보았을 때 확진자의 경우 seasonal(계절성) 그래프, 사망자의 경우 trend(추세) 그래프 와 유사한 패턴을 가지고 있는 것을 알 수 있습니다.
이는 계절성 요소에 대한 예측이 상당히 정확하다는 뜻으로 예측 모델이 시계열 데이터의 계절성을 잘 파악하고 제거했음을 알 수 있습니다. 또한, 큰 변동 없이 비슷한 패턴이 유지된다는 것은 추세와 잔차 요소에 대한 예측도 상당히 정확하다고 볼 수 있습니다.
※ 참고 ※
(1) Data : 시계열 데이터의 실제 관측치를 나타내는 것으로 주어진 시계열 데이터의 원본 패턴과 특성을 살펴볼 수 있습니다. 일반적으로 'data'는 trend(추세), seasonal(계절성), 잔차(residuals)로 분해되며 이 세가지 성분을 시각적으로 파악할 수 있어 시계열 데이터의 동향과 예측에 대한 인사이트를 얻을 수 있습니다.
(2) Trend : 추세 성분을 나타내는 선으로 장기적인 증가 또는 감소를 알 수 있습니다. 일반적으로는 시간이 지남에 따라 일정한 방향으로 증가하거나 감소합니다.
(3) Seasonal : 계절성 성분은 특정한 주기로 반복되는 패턴을 보여주는 것으로 특정 시간이나 주기에 따라 반복 패턴을 가지고 있다면 주기별로 계절성 성분이 나타납니다.
(4) Remainder : 나머지 성분은 설명되지 않는 잔차 혹은 임의의 요인(추세와 계절성을 제외한 나머지 요소들)으로 인한 변동성을 나타내는 것으로 잔차 변동성을 시각화 합니다.
