이번엔 XGBoost에서 XGBRegression방법을 써서 df_train을 가지고 df_test의 코로나 확진자 및 사망자를 예측하려고 한다.
데이터 전처리
데이터 전처리를 하기 위해 네가지 함수를 만드려고 한다.
첫번째 함수
df_train데이터를 확인해보면 Province_State열에 Null값이 포함되어 있다.
그리고 Country_Region과 Province_State는 object 데이터 타입인데, 이를 가지고 바로 XGBoost 학습에 돌릴수는 없다. 이러한 문제를 해결하기 위해 인코딩이라는 방법을 사용해야 한다.
def categoricalToInteger(df):
df.Province_State.fillna('NaN', inplace=True)
oe = OrdinalEncoder()
df[['Province_State', 'Country_Region', 'Continent']] = oe.fit_transform(df.loc[:, ['Province_State', 'Country_Region', 'Continent']])
return df인코더
데이터 인코딩 방법에는 세가지가 있다. Label Encoding, Ordinal Encoding, One-hot Encoding이 이에 포함된다. Encoding은 sklearn 모듈에 포함되어 있다.
Label & Ordinal
Label Encoding과 Ordinal Encoding의 방법은 똑같다. 단지 차이점이 하나가 존재하는데,
Label Encoding은 타겟 변수를 변환하고,Ordinal Encoding은 속성(featrures)을 변환한다는 점이다.
즉 Ordinal Encoding은 2D 데이터(n_samples, n_features)을 convert하고 Label Encoding은 1D 데이터(n_samples,)을 convert한다.
Label Encoding방법을 가지고 설명을 하자면 혈액형을 예로 들 수 있다.
혈액형은 총 네가지 종류가 있다. 'A', 'B', 'O', 'AB'인데 이 문자형을 가지고는 머신러닝 학습을 하기가 힘들다. 그래서 사용하는것이 인코딩을 해서 Interger형으로 변환을 시켜주는 것인데,
from sklearn.preprocessing import LabelEncoder
blood = ['A','O','O','B','AB','A','B']
le = LabelEncoder()
le.fit(blood)
label = le.transform(blood)
label출력 : array([0, 3, 3, 2, 1, 0, 2], dtype=int32)
blood에는 네가지 혈액형이 분포되어 있다. 이를 Label Encoder에 학습시켜 'A' = 0, 'AB' = 2, 'B' = 2, 'O' = 3으로 변환을 시켜주는 것이다. 이렇게 각 혈액형은 숫자형 카테고리로 변환이 되었다. dtype을 보면 int32가 되어있는 것을 볼 수 있을 것이다. 이를 다시 디코딩하려면 inverse_transform()을 해주면 된다.
이 Label Encoding의 문제점은 순서형 변수로 변환이 되기 때문에 머신러닝 학습에서 가중치를 부여하거나 할 때 작은 숫자에 더 부과되거나 하는 등 문제점이 발견될 수 있다. 그래서 이 인코딩 방식은 선형회귀같은 학습에서는 사용해선 안된다.
One-hot
label Encoding의 단점을 보완하기 위해 사용되는 인코더가 One-hot Encoding이다. 이 인코더는 숫자 카테고리컬로 변환해주는 것이 아니라, 벡터 방식으로 변환을 해준다.
| 혈액형 | Label | One-hot |
| :--------: | :--------: | :--------: |
|A | 1 | {1, 0, 0, 0} |
| AB | 2 | {0, 1, 0, 0} |
| B | 3 | {0, 0, 1, 0} |
| O | 4 | {0, 0, 0, 1} |
Label Encoding에서 는 순서형 변수로 변환이 된것이 One-hot에서는 벡터로 표시된 것을 알 수 있다.
코딩으로 보면
blood = np.array(blood)
blood = blood.reshape(-1, 1)
oe= OneHotEncoder()
oe.fit(blood)
oe_lab = oe.transform(blood)
oe_lab.toarray()여기서 One-hot방식에서 주의할 점이 있는데 입력값으로 2차원 데이터가 필요하다는 것이다. 그리고 문자열 값이 숫자형 값으로 변환이 되어야 한다는데, 왠지 모르게 이번에 그냥 문자형값을 넣었는데 되었다.
그래서 원래는 Label Encoding을 먼저 돌린 후 One-hot Encoding을 돌리는게 좋다고 한다.
여기서 2차원 배열로 바꾸기 위해 reshape(-1, 1)을 사용했는데 -1의 이미는 열의 크기인 1을 고정해놓고 자동적으로 행의 길이를 계산해주는 것이다. -1을 쓴다면 굳이 행이나 열의 크기를 계산하지 않아도 된다.

두번째 함수
def create_features(df):
df['day'] = df['Date'].dt.day
df['month'] = df['Date'].dt.month
df['dayofweek'] = df['Date'].dt.dayofweek
df['dayofyear'] = df['Date'].dt.dayofyear
df['quarter'] = df['Date'].dt.quarter
df['weekofyear'] = df['Date'].dt.weekofyear
return df데이터 프레임에 날짜와 관련된 속성을 추가해준다.
세번째 함수
def train_dev_split(df, days):
date = df['Date'].max() - dt.timedelta(days=days)
return df[df['Date'] <= date], df[df['Date'] > date]교차검정을 위해 train데이터를 train 데이터와 validation 데이터로 구분하기 위한 함수같다. 이때 timedelta라는 클래스를 사용하는데 이는 기간을 표현하기 위해 사용된다다.
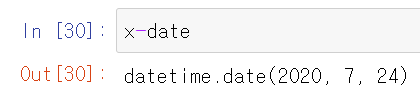
x=dt.date(2020,7,25)
date = dt.timedelta(days=1)
x - datedatetime 변수인 2020/7/25에 날짜 days=1을 뺴주면 2020/7/24일이 된다.
이 방식을 사용하여 사용자가 날짜를 넣고 train데이터에서 가장 최근 날짜에서 해당 날짜만큼을 뺀 값을 이용하여 이보다 작은 값은 train데이터로, 큰 값은 validation값으로 사용하기 위해 반환한다.
네번째 함수
def avoid_data_leakage(df, date=test_date_min):
return df[df['Date'] < date]train 데이터와 test 데이터를 보면 날짜가 겹치는 부분이 존재한다. 그래서 train데이터에서 test의 가장 min한 날짜에 해당하는 데이터를 제거해준다.
위 네가지 함수를 이용해서 train 데이터를 전처리한다.
df_train = avoid_data_leakage(df_train)
df_train = categoricalToInteger(df_train)
df_train = create_features(df_train)
df_train, df_dev = train_dev_split(df_train, 0)그리고 필요한 속성만 따로 빼낸다.
columns = ['day','month','dayofweek','dayofyear','quarter','weekofyear','Province_State', 'Country_Region','Continent','ConfirmedCases','Fatalities']
df_train = df_train[columns]
df_dev = df_dev[columns]test데이터도 마찬가지로 인코더 및 속성을 추가해준다.
df_test['Continent'] = df_test.apply(lambda x: obj.fetch_continent(x['Country_Region']), axis=1)
df_test = categoricalToInteger(df_test)
df_test = create_features(df_test)
columns = ['day','month','dayofweek','dayofyear','quarter','weekofyear','Province_State', 'Country_Region','Continent']
XGBRegression
XGBoost 알고리즘은 앙상블 기법중에 하나이다.
1. 여러 개의 결정 트리를 임의적으로 학습하는 앙상블의 부스팅 유형
2. 순차적 학습 방법. 약한 분류기를 강한 분류기로
3. 분류정확도는 우수하나, Outlier에 약하다.
submission = []
for country in df_train.Country_Region.unique():
df_train1 = df_train[df_train["Country_Region"]==country]
for state in df_train1.Province_State.unique():
df_train2 = df_train1[df_train1["Province_State"]==state]
train = df_train2.values
X_train, Y_train = train[:,:-2], train[:,-2:]
model1 = XGBRegressor(n_estimators=1000)
model1.fit(X_train, Y_train[:,0])
model2 = XGBRegressor(n_estimators=1000)
model2.fit(X_train, Y_train[:,1])
df_test1 = df_test[(df_test["Country_Region"]==country) & (df_test['Province_State']==state)]
ForecastId = df_test1.ForecastId.values
df_test2 = df_test1[columns]
y_pred1 = model1.predict(df_test2.values)
y_pred2 = model2.predict(df_test2.values)
for i in range(len(y_pred1)):
d = {'ForecastId':ForecastId[i], 'ConfirmedCases':y_pred1[i], 'Fatalities':y_pred2[i]}
submission.append(d)
df_submit = pd.DataFrame(submission)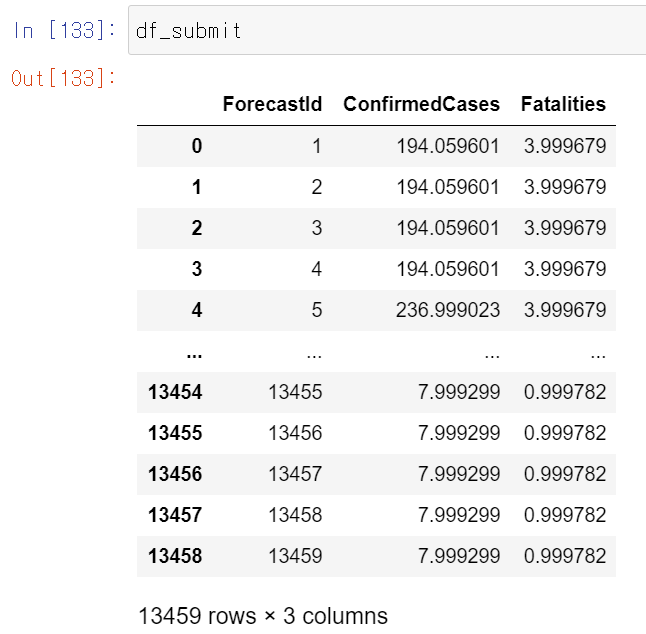
df_forcast = pd.concat([df_test,df_submit.iloc[:,1:]], axis=1)
df_world_f = df_forcast.copy()
df_world_f = df_world_f.groupby('Date',as_index=False)['ConfirmedCases','Fatalities'].sum()
df_world_f = add_daily_measures(df_world_f)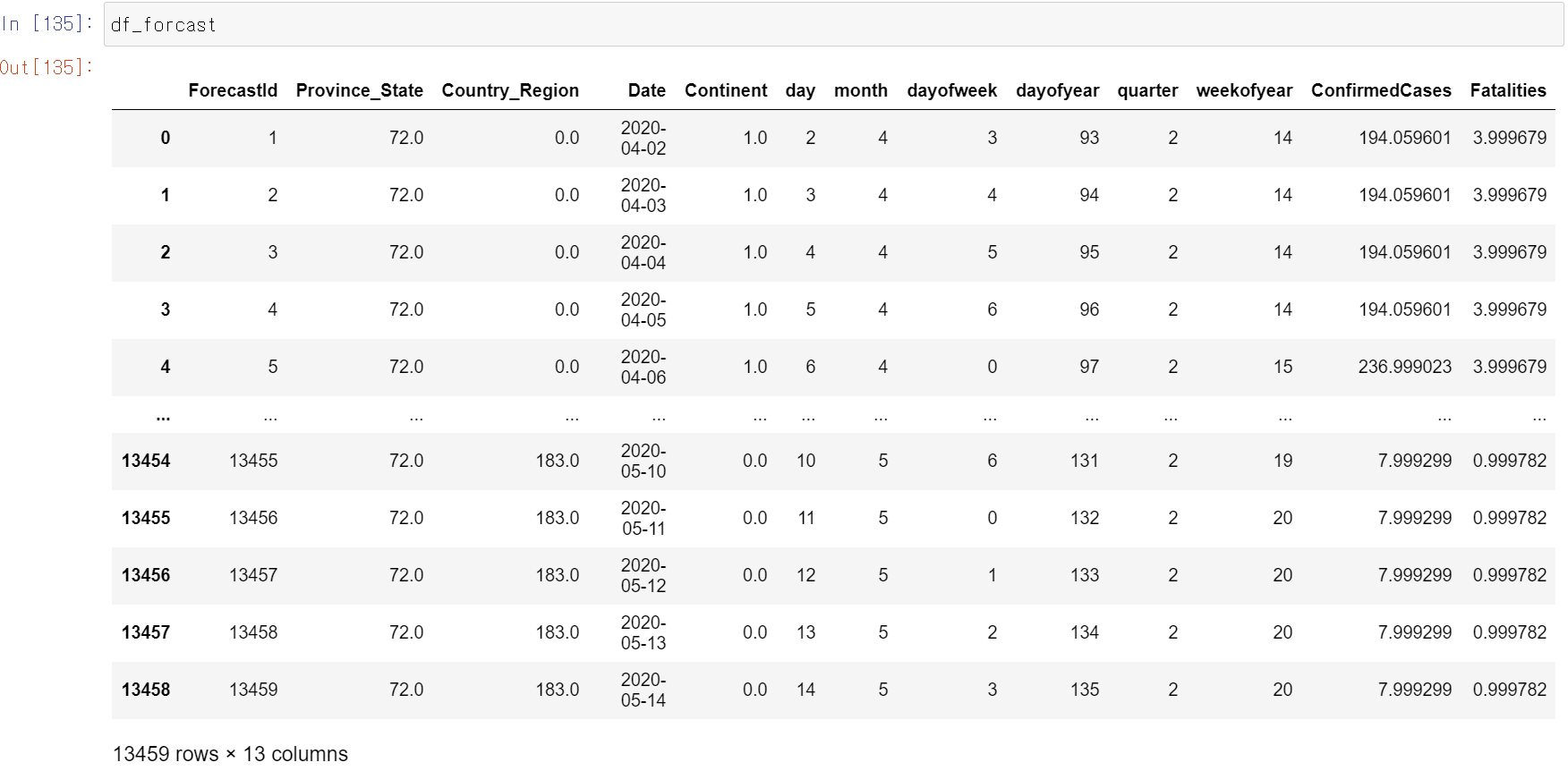
df_world = avoid_data_leakage(df_world)
fig = go.Figure(data=[
go.Bar(name='Total Cases', x=df_world['Date'], y=df_world['ConfirmedCases']),
go.Bar(name='Total Cases Forecasted', x=df_world_f['Date'], y=df_world_f['ConfirmedCases'])
])
# Change the bar mode
fig.update_layout(barmode='group', title='Worldwide Confirmed Cases + Forecasted Cases')
fig.show().png)
