데이터 구성
Pregnancies : 임신 횟수
Glucose : 2시간 동안의 경구 포도당 내성 검사에서 혈장 포도당 농도
BloodPressure : 이완기 혈압 (mm Hg)
SkinThickness : 삼두근 피부 주름 두께 (mm), 체지방을 추정하는데 사용되는 값
Insulin : 2시간 혈청 인슐린 (mu U / ml)
BMI : 체질량 지수 (체중kg / 키(m)^2)
DiabetesPedigreeFunction : 당뇨병 혈통 기능
Age : 나이
Outcome : 768개 중에 268개의 결과 클래스 변수(0 또는 1)는 1이고 나머지는 0입니다.
데이터셋 로드
df = pd.read_csv("http://bit.ly/data-diabetes-csv")
df.shape
=>(768, 9)
df.head(2)
=>
Pregnancies Glucose BloodPressure SkinThickness Insulin BMI DiabetesPedigreeFunction Age Outcome
0 6 148 72 35 0 33.6 0.627 50 1
1 1 85 66 29 0 26.6 0.351 31 0
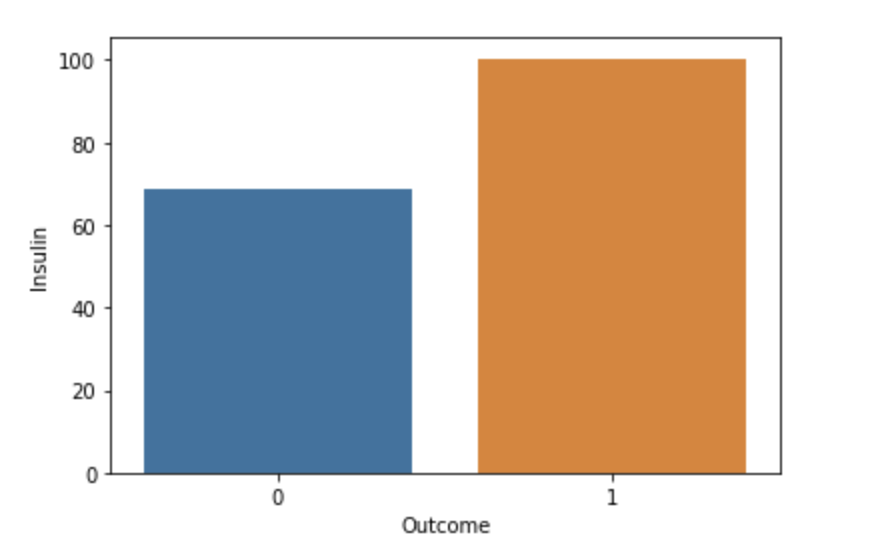
Outcome별 Insulin
sns.barplot(data=df, x="Outcome", y="Insulin", ci=None)
학습, 예측해야 할 값
label_name = "Insulin"
train = df[df[label_name] > 0]
train.shape
=> (394, 9)
test = df[df[label_name] == 0]
test.shape
=> (374, 9)
학습, 예측에 사용할 컬럼
feature_names = df.columns.tolist()
feature_names.remove(label_name)
feature_names
=>
['Pregnancies',
'Glucose',
'BloodPressure',
'SkinThickness',
'BMI',
'DiabetesPedigreeFunction',
'Age',
'Outcome']
학습, 예측 데이터셋 만들기
X_train = train[feature_names]
X_train.shape
=> (394, 8)
y_train = train[label_name]
y_train.shape
=> (394,)
X_test = test[feature_names]
X_test.shape
=> (374, 8)
y_test = test[label_name]
y_test.shape
=> (374,)
머신러닝 알고리즘
from sklearn.ensemble import RandomForestRegressor
model = RandomForestRegressor(n_estimators=100, random_state=42, n_jobs=-1)
model
=> RandomForestRegressor(n_jobs=-1, random_state=42)
학습
model.fit(X_train, y_train)
cross validation 학습 세트의 오차 측정
cross_val_predict 함수 예시
cross_val_predict(
estimator,
X,
y=None,
*,
groups=None,
cv=None,
n_jobs=None,
verbose=0,
fit_params=None,
pre_dispatch='2*n_jobs',
method='predict',
)
from sklearn.model_selection import cross_val_predict
y_predict = cross_val_predict(model, X_train, y_train, cv=5, n_jobs=-1, verbose=2)
y_predict[:5]
=> array([ 42.89, 269.46, 74.45, 335.76, 274.58])
y_train.shape[0]
=> 394
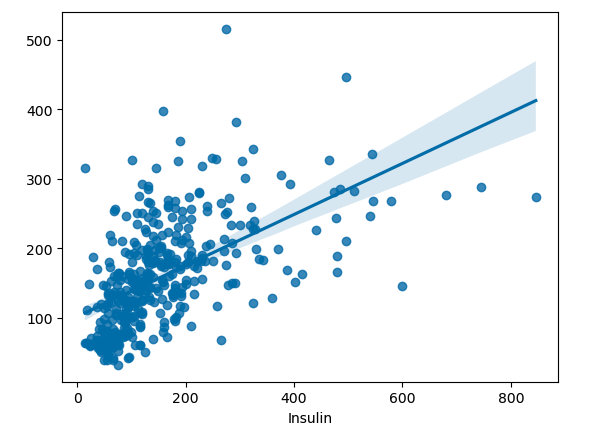
실제값 - 예측값 차이 시각화
sns.regplot(x=y_train, y=y_predict)
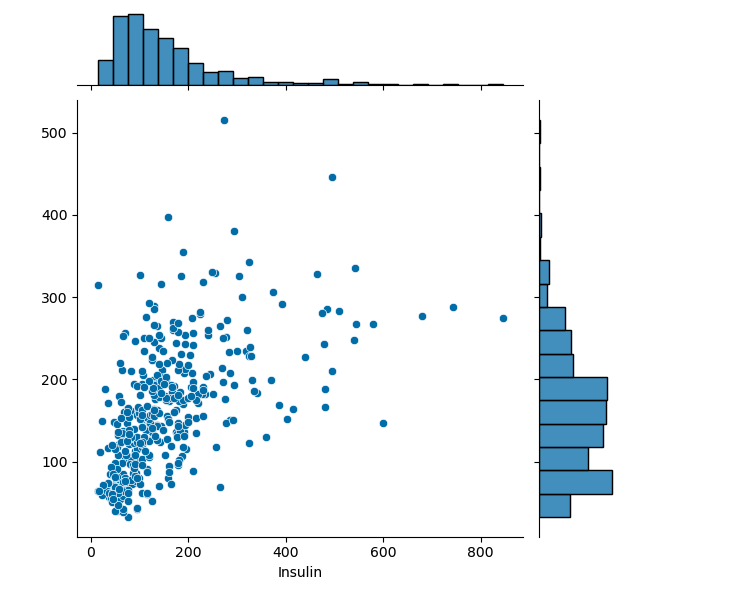
sns.jointplot(x=y_train, y=y_predict)
from sklearn.metrics import r2_score
r2_score(y_train, y_predict)
=>0.3116994764197346
df_y = pd.DataFrame({"true" : y_train, "predict" : y_predict})
df_y

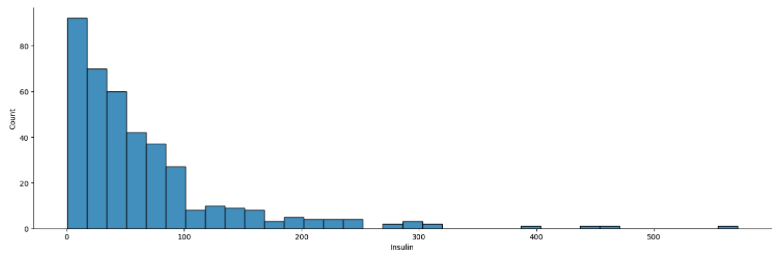
오차 구하기
error = abs(y_train - y_predict)
error.describe()
count 394.000000
mean 65.332944
std 73.695897
min 0.360000
25% 18.702500
50% 44.540000
75% 80.412500
max 571.420000
Name: Insulin, dtype: float64
sns.displot(error, aspect=5)
MAE(Mean Absolute Error)
- 예측값과 실제값의 차이에 대한 절대값의 평균(오차값의 평균)
mae = abs(y_train - y_predict).mean()
mae
=> 65.33294416243655
MAPE(Mean Absolute Percentage Error)
- (실제값 - 예측값 / 실제값)의 절대값에 대한 평균
- 값이 작을수록 예측을 잘 한 것
mape = (abs(y_train - y_predict)/y_train).mean()
mape
=> 0.5851451302268017
MSE(Mean Squared Error)
- 실제값 - 예측값의 차이 제곱의 평균
- MAE와 비슷해 보이나 제곱을 통해 음수를 양수로 변환함
mse = np.square(y_train - y_predict).mean()
mse
=> 9685.694362436549
RMSE(Root Mean Squared Error)
RMSE = np.sqrt(mse)
RMSE
=> 98.41592534969404
트리 알고리즘 분석
피처의 중요도
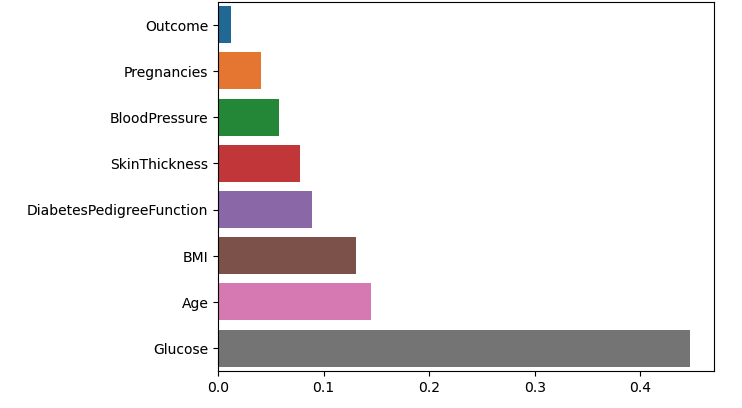
model.feature_importances_
=> array([0.04105672, 0.44740611, 0.05766466, 0.07770453, 0.13022072,
0.0891203 , 0.14501469, 0.01181228])
fi - pd.Series(model.feature_importances_)
fi.index = model.feature_names_in_
fi = fi.sort_values()
sns.barplot(x=fi, y=fi.index);
예측
y_test_pred = model.predict(X_test)
y_test_pred[:5]
=> array([175.33, 49.08, 275.41, 151.23, 184.23])
실제값과 예측값 비교
train.groupby("Outcome")["Insulin"].describe()
count mean std min 25% 50% 75% max
Outcome
0 264.0 130.287879 102.482237 15.0 66.0 102.5 161.25 744.0
1 130.0 206.846154 132.699898 14.0 127.5 169.5 239.25 846.0
test["Insulin"] = y_test_pred
test.groupby('Outcome')['Insulin'].describe()
count mean std min 25% 50% 75% max
Outcome
0 236.0 148.920847 68.982874 46.45 92.340 138.25 192.1050 466.61
1 138.0 218.066522 83.522211 67.21 162.175 209.53 259.8225 624.54






