EDA
탐색적 데이터 분석, Exploratory Data Analysis
데이터 입출력
Colab 세팅
- 아래 코드 실행 후, google 계정 확인 창이 나타납니다.
- 성공적으로 단계 처리하면, 구글 드라이브가 연결됨.
from google.colab import drive
drive.mount('/content/drive')데이터 입력
pd.read_csv('filepath', sep=',', na_values='NA', encoding='utf-8')pd.read_excel('filepath', sheet_name='Sheet1')pd.read_json('filepath')
데이터 출력
df.to_csv('filepath', sep=',', header=False, index=False, encoding='utf-8')df.to_excel('filepath', sheet_name='Output')df.to_json('filepath', indent=2)
DataFrame 기본
df.head(10)df.tail(10)
모든 행/열 출력하게 하기
pd.set_option('display.max_columns', None)
display.max_columns: 20 (Default)pd.set_option('display.max_rows', None)
display.max_rows: 60 (Default)
DataFrame 요약/통계 정보 확인하기
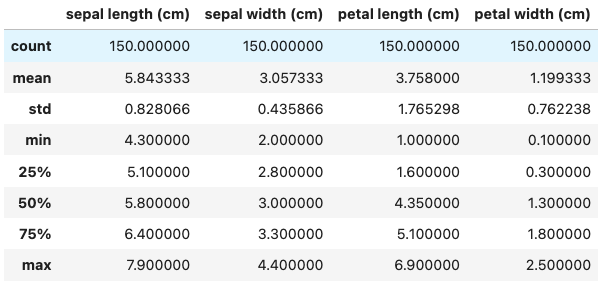
df.info(): Print a concise summary of a DataFrame.<class 'pandas.core.frame.DataFrame'> RangeIndex: 150 entries, 0 to 149 Data columns (total 4 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 sepal length (cm) 150 non-null float64 1 sepal width (cm) 150 non-null float64 2 petal length (cm) 150 non-null float64 3 petal width (cm) 150 non-null float64 dtypes: float64(4) memory usage: 4.8 KBdf.describe(): Return Series or DataFrame having Summary statistics of the Series or Dataframe provided.
데이터 변환
- 데이터 Setting
sample_size = 1000
arr1 = 5 * np.random.randn(sample_size) + 53.9
arr2 = 4 * np.random.randn(sample_size) + 32.7표준 정규화 (Z-표준화)
- numpy, series, dataframe 함수 이용
(arr - np.mean(arr)) / np.std(arr)df["z-score"] = (df["column"] - df["column"].mean()) / df["column"].std()
- scipy.stats.zscore 함수 이용
- `scipy.stats.zscore(arr)`
- sklearn.preprocessing.StandardScaler 클래스 함수 이용
- `sklearn.preprocessing.StandardScaler().fit_transform(array_like)`
Way 1. numpy, pandas 함수를 이용해서 표준화
df = pd.DataFrame({"data1": arr1, "data2": arr2})
mean1 = np.mean(df["data1"])
mean2 = np.mean(df["data2"])
std1 = np.std(df["data1"], ddof=1)
std2 = np.std(df["data2"], ddof=1)
df["data1_z_score"] = (df["data1"] - mean1) / std1
df["data2_z_score"] = (df["data2"] - mean2) / std2
dfdf = pd.DataFrame({"data1": arr1, "data2": arr2})
mean1, mean2 = tuple(df.mean().to_list())
std1, std2 = tuple(df.std().to_list())
df["data1_z_score"] = (df["data1"] - mean1) / std1
df["data2_z_score"] = (df["data2"] - mean2) / std2
df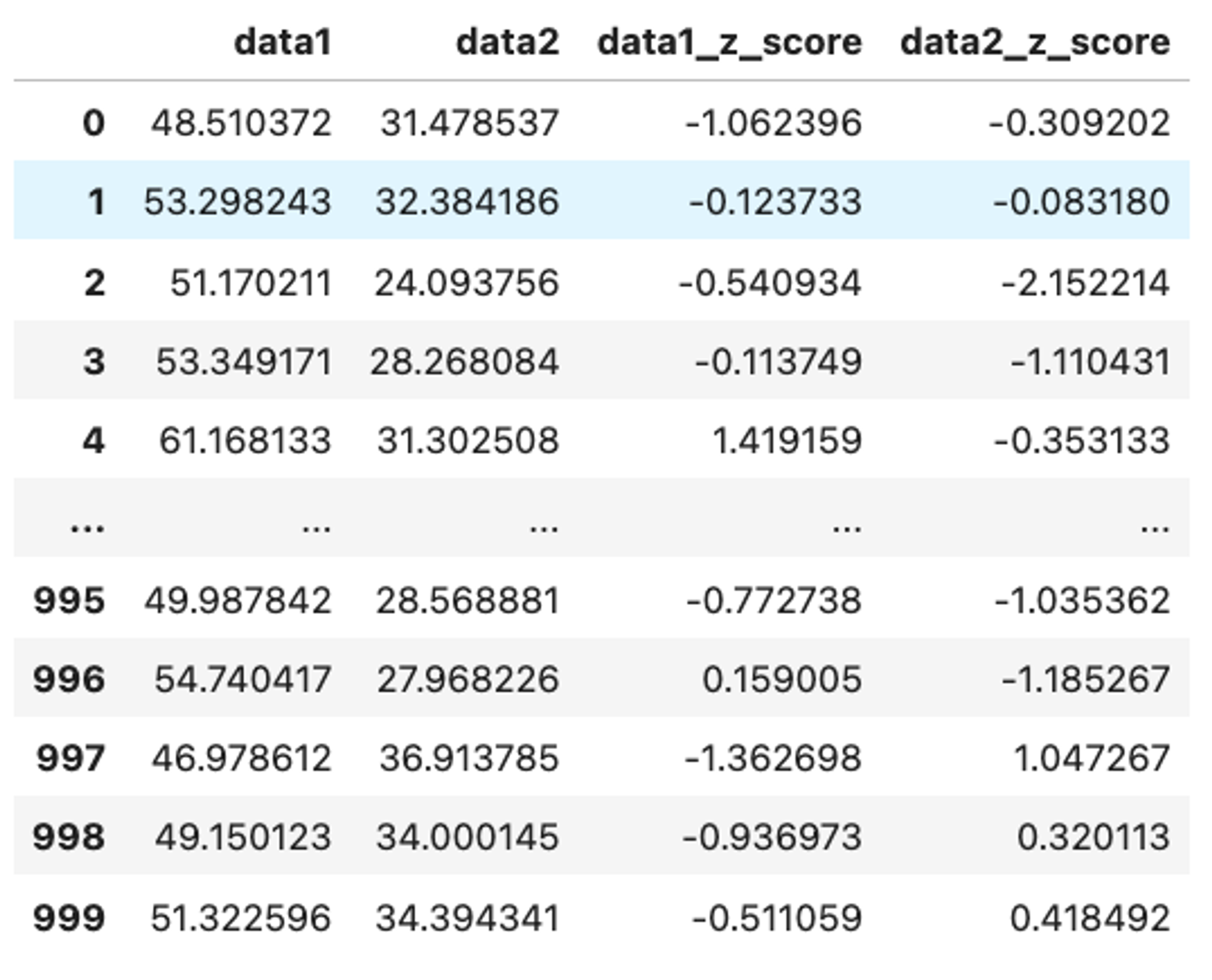
Way 2. scipy.stats 모듈의 zscore 함수를 사용해서 표준화
import scipy.stats as stats
df = pd.DataFrame({"data1": arr1, "data2": arr2})
df["data1_z_score"] = stats.zscore(df["data1"], ddof=1)
df["data2_z_score"] = stats.zscore(df["data2"], ddof=1)
dfWay 3. sklearn.preprocessing 모듈의 StandardScaler 클래스 사용해서 표준화
from sklearn.preprocessing import StandardScaler
df = pd.DataFrame({"data1": arr1, "data2": arr2})
standard_scaler = StandardScaler()
df_z_score = pd.DataFrame(standard_scaler.fit_transform(df), columns=["data1_z_score", "data2_z_score"])
# 1. pd.concat([df1, df2])
df_result1 = pd.concat([df, df_z_score], axis=1)
# 2. df.join(df2, how="left")
df_result3 = df.join(df_z_score)Min-Max 정규화
- numpy, series, dataframe 함수 이용
(arr - np.mean(arr)) / np.std(arr)df["z-score"] = (df["column"] - df["column"].mean()) / df["column"].std()
- sklearn.preprocessing.MinMaxScaler 클래스 함수 이용
- `sklearn.preprocessing.MinMaxScaler().fit_transform(array_like)`
Way 1. numpy, pandas 함수를 이용해서 표준화
df = pd.DataFrame({"data1": arr1, "data2": arr2})
max1 = np.max(df["data1"])
max2 = np.max(df["data2"])
min1 = np.min(df["data1"])
min2 = np.min(df["data2"])
df["data1_min_max_score"] = (df["data1"] - min1) / (max1 - min1)
df["data2_min_max_score"] = (df["data2"] - min2) / (max2 - min2)
dfdf = pd.DataFrame({"data1": arr1, "data2": arr2})
max1, max2 = tuple(df.max().to_list())
min1, min2 = tuple(df.min().to_list())
df["data1_min_max_score"] = (df["data1"] - min1) / (max1 - min1)
df["data2_min_max_score"] = (df["data2"] - min2) / (max2 - min2)
dfWay 2. sklearn.preprocessing 모듈의 MinMaxScaler 클래스 사용해서 표준화
from sklearn.preprocessing import MinMaxScaler
df = pd.DataFrame({"data1": arr1, "data2": arr2})
min_max_scaler = MinMaxScaler()
df_min_max_score = pd.DataFrame(min_max_scaler.fit_transform(df), columns=["data1_min_max_score", "data2_min_max_score"])
df.join(df_min_max_score)정규분포변환
- skewness (왜도)에 따라 기존의 데이터의 분포형태를 변경해야한다.
- 독립변수 값이 증가함에 따라 종속변수가 더 빠르게 증가할 경우,
- 로그 변환을 시도하는것이 좋다.
- 로그 변환시 모든 값을 양수로 만들어야 한다.
- 독립변수가 값이 증가함에 따라 종속변수가 더 빠르게 감소할 경우,
- 제곱변환을 시도하는것이 좋다.
- `scipy.stats.skew(arr)`
왜도 (Skewness) 제곱근 변환 로그 변환 역수 변환 양수 sqrt(x) log(x) log10(x) 1 / x 음수 sqrt(max(x + 1) - x) log(max(x + 1) - x) log10(max(x + 1) - x) 1 / (max(x + 1) - x)
범주화(Categorization), 이산형화(Discretization)
연속형 변수를 범주형 변수로 변환하는 작업이다.
데이터 Setting
def makeScore(x):
result = round(x)
if result > 100:
return 100
elif result < 50:
return 50
else:
return result
data = list(map(lambda x: makeScore(x), np.random.randn(1000) * 12 + 70))
df = pd.DataFrame(data, columns=["math_score"])
import matplotlib.pyplot as plt
plt.hist(df, bins=5, range=[50,100], rwidth=0.9)
plt.show()way1. loc함수와 조건을 사용해서 구간을 직접 설정
- 조건을 사용해서, 값을 변경하고자 할때
loc을 통해서 변경해야한다.
df["grade"] = np.nan
df["grade"] = df["grade"].astype("str")
# df[df["math_score"] >= 90]["grade"] = "A" # SettingWithCopyWarning
df.loc[df["math_score"] >= 90, "grade"] = "A"
df.loc[(df["math_score"] >= 80) & (df["math_score"] < 90), "grade"] = "B"
df.loc[(df["math_score"] >= 70) & (df["math_score"] < 80), "grade"] = "C"
df.loc[(df["math_score"] >= 60) & (df["math_score"] < 70), "grade"] = "D"
df.loc[df["math_score"] < 60, "grade"] = "F"
dfway2. pandas.cut 함수 사용
df["grade2"] = pd.cut(df["math_score"], bins=[0, 60, 70, 80, 90, 101], labels= ["F", "D", "C", "B", "A"], right=False, include_lowest=True)
# cut 함수
pd.cut(
x,
bins,
right: 'bool' = True,
labels=None,
retbins: 'bool' = False,
precision: 'int' = 3,
include_lowest: 'bool' = False,
duplicates: 'str' = 'raise',
ordered: 'bool' = True,
)way3. pandas.qcut 함수 사용
- cut() 함수는
bins를 직접 수치로 지정하는 반면,- qcut() 함수는 나누고자 하는 범주 개수(
q)를 정해주면,- 각 범주에 균등한 수의 데이터가 채워지도록 범주를 나눈다.
- qcut() 함수는 나누고자 하는 범주 개수(
df["grade3"] = pd.qcut(df["math_score"], q=5, labels=["F", "D", "C", "B", "A"])
# qcut 함수
pd.qcut(
x,
q,
labels=None,
retbins: 'bool' = False,
precision: 'int' = 3,
duplicates: 'str' = 'raise',
)PCA (Principal Component Analysis, 주성분 분석)
주성분 분석이란 여러 변수들의 변량을 주성분이라고 불리는, 서로 상관성이 높은 여러 변수들의 선형조합으로 만든 새로운 변수들로 요약, 축소하는 기법이다.
sklearn.decomposition.PCA클래스 활용
sklearn.decomposition.PCA(
n_components=None,
*,
copy=True,
whiten=False,
svd_solver='auto',
tol=0.0,
iterated_power='auto',
n_oversamples=10,
power_iteration_normalizer='auto',
random_state=None,
)df = pd.read_csv("https://raw.githubusercontent.com/YoungjinBD/dataset/main/iris.csv")
df = df.drop(columns=["species"])
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
# 표준 정규화
standard_scaler = StandardScaler()
df = pd.DataFrame(standard_scaler.fit_transform(df), columns=df.columns)
# PCA 수행
pca = PCA(n_components=4)
p_score = pca.fit_transform(df)
print(pca.explained_variance_ratio_)
# 출력
# [0.72962445 0.22850762 0.03668922 0.00517871]필터링 (Filtering)
df[df['column'] == 1]- bool 타입의 Series 혹은 ndarray 를 통해 필터링을 수행할 수 있다.
assert (df[np.isin(df.season, [1, 2])].index == df[(df.season == 1) | (df.season == 2)].index).all()
assert (df[np.isin(df.season, [1, 2])].index == df.iloc[np.isin(df.season, [1, 2])].index).all()
assert (df[np.isin(df.season, [1, 2])].index == df.loc[np.isin(df.season, [1, 2])].index).all()
assert (df[df.season.isin([1, 2])].index == df[np.isin(df.season, [1, 2])].index).all()df.isin(array_like)
df.isin(values: 'Series | DataFrame | Sequence | Mapping') -> 'DataFrame'np.isin(array_like1, array_like2)
np.isin(arr1, arr2)np.in1d(arr1, arr2)
np.isin(
element,
test_elements,
assume_unique=False,
invert=False,
*,
kind=None,
) -> ndarrayOutlier 처리 (이상치 처리) - quantile
df.quantile(.45, numeric_only=True)df.quantile([.01, .99], numeric_only=True)
df.quantile(
q: 'float | AnyArrayLike | Sequence[float]' = 0.5,
axis: 'Axis' = 0,
numeric_only: 'bool' = False,
interpolation: 'QuantileInterpolation' = 'linear',
method: "Literal['single', 'table']" = 'single',
) -> 'Series | DataFrame'
percentile_1, percentile_99 = tuple(df['casual'].quantile([0.01, 0.99]).values)
cond_1 = df['casual'] >= percentile_1
cond_2 = df['casual'] <= percentile_99
df[cond_1 & cond_2]결측치 파악 (isna)
df.isna().sum()df.isnull()==df.isna()df.notnull()==df.notna()
결측치가 하나라도 존재하는 행 개수
df.isna().any(axis=1).sum()
결측치가 하나라도 존재하는 인덱스 목록
df[df.isna().any(axis=1)].index.tolist()
결측치 치환 (fillna)
df['column'].fillna(value)- 결측치를 지정값으로 치환
df['column'].fillna(method='ffill', limit=10)- 결측치를 인접한 값으로 대체
- 결측치가 있는 행의 직전 행에 있는 값으로 대체
df['column'].fillna(method='bfill')- 결측치를 인접한 값으로 대체
- 결측치가 있는 행의 바로 다음 행에 있는 값으로 대체
df['column'].replace(oldValue, newValue)
df.fillna(
value: 'Hashable | Mapping | Series | DataFrame | None' = None,
*,
method: 'FillnaOptions | None' = None,
axis: 'Axis | None' = None,
inplace: 'bool_t' = False,
limit: 'int | None' = None,
downcast: 'dict | None | lib.NoDefault' = <no_default>,
) -> 'Self | None'결측치를 그룹별 평균값으로 대체
- groupby(), transform() 함수 활용
df.groupby("column1")["column2"].transform("mean")
df = pd.read_csv("https://raw.githubusercontent.com/YoungjinBD/dataset/main/titanic.csv")
df_groupby_age_mean = df.groupby(by="Pclass")["Age"].transform("mean")
df["Age"] = df["Age"].fillna(df_groupby_age_mean)
assert df["Age"].isna().sum() == 0
df.tail()결측치 제거 (dropna)
df.dropna()df.dropna(how='any', axis=0)df.dropna(how='all')
df.dropna(
*,
axis: 'Axis' = 0,
how: 'AnyAll | lib.NoDefault' = <no_default>,
thresh: 'int | lib.NoDefault' = <no_default>,
subset: 'IndexLabel | None' = None,
inplace: 'bool' = False,
ignore_index: 'bool' = False,
) -> 'DataFrame | None'추가 함수 (df.isin)
df.isin(['A', 'B'])df['column'].isin([1, 2, 3])
df.isin(values: 'Series | DataFrame | Sequence | Mapping') -> 'DataFrame'정렬과 reset_index
df_new.sort_values(by = ['Petal.Length', 'Petal.Width']).reset_index(drop=True)
df.sort_values, df.sort_index
df.sort_values('column')df.sort_values(['column1', 'column2'], ascending=False)
df.sort_values(
by: 'IndexLabel',
*,
axis: 'Axis' = 0,
ascending: 'bool | list[bool] | tuple[bool, ...]' = True,
inplace: 'bool' = False,
kind: 'SortKind' = 'quicksort',
na_position: 'str' = 'last',
ignore_index: 'bool' = False,
key: 'ValueKeyFunc | None' = None,
) -> 'DataFrame | None'
df.reset_index
df.reset_index(drop=True)
df.reset_index(
level: 'IndexLabel | None' = None,
*,
drop: 'bool' = False,
inplace: 'bool' = False,
col_level: 'Hashable' = 0,
col_fill: 'Hashable' = '',
allow_duplicates: 'bool | lib.NoDefault' = <no_default>,
names: 'Hashable | Sequence[Hashable] | None' = None,
) -> 'DataFrame | None'
임의 추출 (sample)
df.sample(n = 10).reset_index(drop=False)df.sample(frac=0.1, replace=True).reset_index()
df.sample(
n: 'int | None' = None,
frac: 'float | None' = None,
replace: 'bool_t' = False,
weights=None,
random_state: 'RandomState | None' = None,
axis: 'Axis | None' = None,
ignore_index: 'bool_t' = False,
) -> 'Self'
색인 (reset_index, set_index)
df.sample(n = 10).reset_index(drop=False)df.sample(frac=0.1, replace=True).reset_index()df[['column1', 'column2', 'column3']].set_index(['column1', 'column2'])df.xs(key='index1_value')df.xs(key=('index1_value', 'index2_value')
df.reset_index(
level: 'IndexLabel | None' = None,
*,
drop: 'bool' = False,
inplace: 'bool' = False,
col_level: 'Hashable' = 0,
col_fill: 'Hashable' = '',
allow_duplicates: 'bool | lib.NoDefault' = <no_default>,
names: 'Hashable | Sequence[Hashable] | None' = None,
) -> 'DataFrame | None'
df.set_index(
keys,
*,
drop: 'bool' = True,
append: 'bool' = False,
inplace: 'bool' = False,
verify_integrity: 'bool' = False,
) -> 'DataFrame | None'
# cross section
df2.xs(
key: 'IndexLabel',
axis: 'Axis' = 0,
level: 'IndexLabel | None' = None,
drop_level: 'bool_t' = True,
) -> 'Self'데이터 타입 변환 (df.astype)
df['season'].astype('str')df['holiday'].astype('bool')
df.astype(
dtype,
copy: 'bool_t | None' = None,
errors: 'IgnoreRaise' = 'raise',
) -> 'Self'
Label Encoding
- 카테고리 컬럼의 값을 코드형 숫자 값으로 변환한다.
sklearn.preprocessor.LabelEncoder클래스 활용
from sklearn.preprocessing import LabelEncoder
labelEncoder = LabelEncoder()
# fit, transform
labelEncoder.fit(sorted(np.unique(df["Sex"])))
df["Sex"] = labelEncoder.transform(df["Sex"])
# fit_transform
df["Embarked"] = labelEncoder.fit_transform(df["Embarked"])One-Hot Encoding
1. OneHotEncoder (sklearn.preprocessing)
OneHotEncoder(
*,
categories='auto',
drop=None,
sparse_output=True,
dtype=<class 'numpy.float64'>,
handle_unknown='error',
min_frequency=None,
max_categories=None,
feature_name_combiner='concat',
)- 장점
- 통합된 머신러닝 파이프라인:
OneHotEncoder는scikit-learn의 다른 도구들과 통합되어 머신러닝 파이프라인에 쉽게 포함될 수 있습니다. - 다양한 옵션 제공: 다양한 인코딩 옵션을 제공하며, 범주형 특징의 처리 방식을 세부적으로 조정할 수 있습니다.
- 자동 처리: 새로운 데이터에 대해 자동으로 인코딩을 적용할 수 있으며, 훈련 데이터와 동일한 방식으로 인코딩합니다.
- 통합된 머신러닝 파이프라인:
- 단점
- 상대적으로 복잡한 사용법:
fit과transform메서드를 사용해야 하므로, 사용법이 약간 더 복잡할 수 있습니다.
- 상대적으로 복잡한 사용법:
import pandas as pd
from sklearn.preprocessing import OneHotEncoder
# 예제 데이터 생성
df = pd.DataFrame({
'color': ['red', 'blue', 'green', 'blue', 'red']
})
# OneHotEncoder 사용
encoder = OneHotEncoder(sparse_output=False)
encoded = encoder.fit_transform(df[['color']])
encoded_df = pd.DataFrame(encoded, columns=encoder.get_feature_names_out(['color']))
print(encoded_df)sparse_output 옵션
True: 원-핫 인코딩 결과를 희소 행렬로 반환합니다. 메모리 효율성이 높습니다.False: 원-핫 인코딩 결과를 밀집 행렬로 반환합니다. 메모리 사용량이 더 많지만, 일부 상황에서는 더 편리할 수 있습니다.
sparse_output 옵션은 scikit-learn의 OneHotEncoder에서 희소 행렬(sparse matrix)과 밀집 행렬(dense matrix) 중 어떤 형태로 출력을 받을지를 결정하는 옵션입니다.
희소 행렬 (Sparse Matrix)
- 희소 행렬은 대부분의 요소가 0인 행렬로, 메모리 효율성을 높이기 위해 0이 아닌 요소들만 저장하는 방식입니다.
- 특히 원-핫 인코딩 결과는 많은 0값을 포함하게 되므로,
- 희소 행렬을 사용하면 메모리 사용을 줄일 수 있습니다.
밀집 행렬 (Dense Matrix)
- 밀집 행렬은 모든 요소를 저장하는 일반적인 행렬 형식입니다.
- 메모리 사용량은 더 많지만, 계산이 간단하고 일부 알고리즘에서는 밀집 행렬을 필요로 할 수 있습니다.
import pandas as pd
from sklearn.preprocessing import OneHotEncoder
# 예제 데이터 생성
df = pd.DataFrame({
'color': ['red', 'blue', 'green', 'blue', 'red']
})
# OneHotEncoder 사용 (희소 행렬 출력)
encoder_sparse = OneHotEncoder(sparse_output=True)
encoded_sparse = encoder_sparse.fit_transform(df[['color']])
print("Sparse Matrix:\n", encoded_sparse)
# OneHotEncoder 사용 (밀집 행렬 출력)
encoder_dense = OneHotEncoder(sparse_output=False)
encoded_dense = encoder_dense.fit_transform(df[['color']])
encoded_df_dense = pd.DataFrame(encoded_dense, columns=encoder_dense.get_feature_names_out(['color']))
print("\nDense Matrix:\n", encoded_df_dense)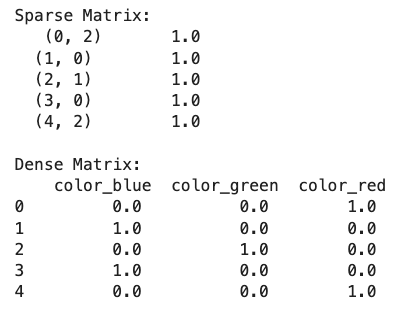
2. pd.get_dummies
pd.get_dummies(
data,
prefix=None,
prefix_sep: 'str | Iterable[str] | dict[str, str]' = '_',
dummy_na: 'bool' = False,
columns=None,
sparse: 'bool' = False,
drop_first: 'bool' = False,
dtype: 'NpDtype | None' = None,
) -> 'DataFrame'- 장점
- 간편함: 한 줄의 코드로 쉽게 원-핫 인코딩을 수행할 수 있습니다.
- DataFrame 통합: 인코딩된 데이터를 바로
DataFrame에 포함할 수 있으며, 원본 데이터와 쉽게 결합할 수 있습니다.
- 단점
- 자동화 부족: 새로운 데이터에 대해 자동으로 인코딩을 적용하는 기능이 없습니다.
- 기능 제한:
OneHotEncoder보다 제공되는 옵션이 적습니다.
python코드 복사
import pandas as pd
# 예제 데이터 생성
df = pd.DataFrame({
'color': ['red', 'blue', 'green', 'blue', 'red']
})
# get_dummies 사용
encoded_df = pd.get_dummies(df, columns=['color'])
print(encoded_df)
비교 요약
| 특성 | OneHotEncoder (sklearn) | get_dummies (pandas) |
|---|---|---|
| 통합된 머신러닝 파이프라인 | 예, scikit-learn 파이프라인 통합 | 아니요 |
| 사용법 | 상대적으로 복잡 (fit, transform) | 간단 (한 줄 코드) |
| 자동 처리 | 예, 새로운 데이터에 대해 자동 처리 | 아니요 |
| 제공 옵션 | 다양 (다양한 인코딩 옵션 제공) | 제한적 (기본 원-핫 인코딩 제공) |
| 결과 형태 | numpy 배열 (또는 DataFrame) | DataFrame |
Transaction Encoding
mlxtend.preprocessing.TransactionEncoder
import pandas as pd
from mlxtend.preprocessing import TransactionEncoder
# 예제 거래 데이터
transactions = [
['milk', 'bread', 'eggs'],
['bread', 'butter'],
['milk', 'bread', 'butter'],
['bread', 'eggs']
]
# TransactionEncoder 인스턴스 생성
te = TransactionEncoder()
# 거래 데이터를 One-Hot 인코딩 형식으로 변환
te_ary = te.fit(transactions).transform(transactions)
# 변환된 데이터를 데이터프레임으로 변환
df = pd.DataFrame(te_ary, columns=te.columns_)
print(df)
# 출력 결과
bread butter eggs milk
0 True False True True
1 True True False False
2 True True False True
3 True False True False평활화 (Smoothing)
평활화의 목적은 값의 변화를 매끄럽게 하여 데이터의 추세(경향)를 알기 쉽게 하는 것이다.
이동평균 (MA, Moving Average)
이전 n개의 데이터의 비가중 / 가중 평균
- 단순이동평균 (Simple Moving Average)
```
df.rolling(
window: 'int | dt.timedelta | str | BaseOffset | BaseIndexer',
min_periods: 'int | None' = None,
center: 'bool_t' = False,
win_type: 'str | None' = None,
on: 'str | None' = None,
axis: 'Axis | lib.NoDefault' = <no_default>,
closed: 'IntervalClosedType | None' = None,
step: 'int | None' = None,
method: 'str' = 'single',
) -> 'Window | Rolling'
```- 누적이동평균
- 가중이동평균

