딥러닝 기본기 다지기
Chap 10. 딥러닝 모델 설계하기
model = Sequential()
케라스를 통해 입력층, 은닉층, 출력층을 만드는 구문
modle = Sequential()
model.add(Dense(30, input_dim = 16, activation = 'relu'))
model.add(Dense(1, activation = 'sigmoid'))마지막 층은 출력층 담당, 나머지는 모두 은닉층 담당
→ 입력층은 첫번째 Dense() 함수 내의 input_dim 값으로 설정 가능.
Dense()의 첫 번째 인자는 Node의 개수
model.compile()
- 오차함수
새로운 사실 : velog는 span이 적용이 안된다!
| 평균 제곱 계열 (선형 회귀 모델) |
mean_squared_error | 평균 제곱 오차 계산 : mean(square(y_t-y_o) |
|---|---|---|
| 평균 제곱 계열 (선형 회귀 모델) |
mean_absolute_error | 평균 절대 오차 계산 : mean(abs(y_t-y_o) |
| 평균 제곱 계열 (선형 회귀 모델) |
mean_absolute_percentage_error | 평균 절대 백분율 오차 계산 : mean(abs(y_t-y_o)/abs(y_t) |
| 평균 제곱 계열 (선형 회귀 모델) |
mean_squared_logarithmic_error | 평균 제곱 로그 오차 계산 : mean(square((log(y_0) + 1) - (log(y_t) + 1))) |
| 교차 엔트로피 계열 (다항 분류, 이항 분류) |
categorical_crossentropy | 범주형 교차 엔트로피 |
| 교차 엔트로피 계열 (다항 분류, 이항 분류) |
binary_crossentropy | 이항 교차 엔트로피 |
-
optimizer : adam 많이 씀
-
metrics(): 수행 결과를 지정- accuracy, loss, val_acc, val_loss 등 -> 14장
model.fit()
epoch: 반복 횟수atch_size: 한 번에 처리할 속성 개수
Chap 11. 데이터 다루기
- csv : comma separated values
- header : csv 파일 맨 처음에 나오는 데이터를 설명하는 한 줄
- pima-indians-diabetes3.csv
새로운 사실 : velog는 정렬도 안된다!
| pregnant | plasma | pressure | thickness | insulin | bmi | pedigree | age | diabetes | |
|---|---|---|---|---|---|---|---|---|---|
| 0 | 6 | 148 | 72 | 35 | 0 | 33.6 | 0.627 | 50 | 1 |
| 1 | 1 | 85 | 66 | 29 | 0 | 26.6 | 0.351 | 31 | 0 |
| 2 | 8 | 183 | 64 | 0 | 0 | 23.3 | 0.627 | 32 | 1 |
- 샘플 수 : 768개
- 속성 : 8개
- 정보 1(pregnant) : 과거 임신 횟수
- 정보 2(plasma) : 포도당 부하 검사 2시간 후 공복 혈당 농도(mm Hg)
- 정보 3(pressure) : 확장기 혈압(mm Hg)
- 정보 4(thickness) : 삼두근 피부 주름 두께(mm)
- 정보 5(insulin) : 혈청 인슐린(2-hour, mu U/ml)
- 정보 6(BMI) : 체질량 지수(BMI, weight in kg/(height in m)²)
- 정보 7(pedigree) : 당뇨병 가족력
- 정보 8(age) : 나이
- 클래스 : 당뇨(1), 당뇨 아님(0)
Python
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
df = pd.read_csv('./data/pima-indians-diabetes3.csv')
df.head(5)
df["diabetes"].value_counts()
df.describe()
df.corr()
colormap = plt.cm.gist_heat
plt.figure(figsize=(12,12))
sns.heatmap(df.corr(),linewidths=0.1,vmax=0.5, cmap=colormap, linecolor='white', annot=True)
plt.show()
plt.hist(x=[df.plasma[df.diabetes==0], df.plasma[df.diabetes==1]], bins=30, histtype='barstacked', label=['normal','diabetes'])
plt.legend()
plt.hist(x=[df.bmi[df.diabetes==0], df.bmi[df.diabetes==1]], bins=30, histtype='barstacked', label=['normal','diabetes'])
plt.legend()
describe(): 정보별 count(샘플 수), mean(평균), std(표준편차), min(최솟값), 백분위, max(최댓값)
corr(): 각 항목 별 상관관계
seaborn라이브러리를 이용한 그래프
colormap = plt.cm.gist_heat: 색상 구성 결정 matplotlib.org
plt.figure(figsize=(12,12)): 그래프 크기 결정
heatmap: 두 항목씩 짝을 지은 후 각각 어떤 패턴으로 변화하는지 관찰하는 함수 비슷한 패턴으로 변할수록 1에 가까운 값을 출력. 전혀 다르면 0
plt.hist(x, bins, histtype, label)x : 가져올 칼럼들
bins : x축을 몇 개의 막대로 쪼개어 보여 줄 것인지 정하는 변수
baarstacked : 겹친거
label : x 순서대로 이름
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense
import pandas as pd
df = pd.read_csv('./data/pima-indians-diabetes3.csv')
X = df.iloc[:,0:8]
y = df.iloc[:,8]
model = Sequential()
model.add(Dense(12, input_dim=8, activation='relu', name='Dense_1'))
model.add(Dense(8, activation='relu', name='Dense_2'))
model.add(Dense(1, activation='sigmoid',name='Dense_3'))
model.summary()
model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['accuracy'])
history=model.fit(X, y, epochs=100, batch_size=5)
pandas.iloc: 범위만큼 가져와 저장
model
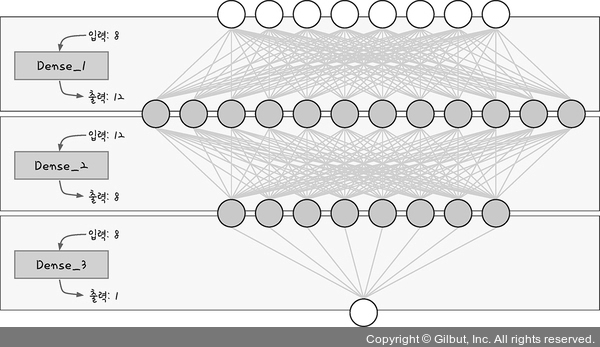
- 입력값 8개
- 은닉층 3개
- 첫번째 은닉층 출력 노드 갯수 12개, 두번째 은닉층 출력 노드 갯수 8개
- 출력값 1개
- 첫번째, 두번째 은닉층 활성화 함수 : relu, 마지막 은닉층 활서오하 함수 : sigmoid
model.summary()실행 결과Model: "sequential"
_________________________________________________________________
Layer (type) | Output Shape | Param #
========================================`Dense_1 (Dense) | (None, 12) | 108
Dense_2 (Dense) | (None, 8) | 104
Dense_3 (Dense) | (None, 1) | 9
========================================
Total params: 221
Trainable params: 221
Non-trainable params: 0
_________________________________________________________________Layer : 층 이름과 유형
Output Shape : 각 층에서 발생하는 출력 개수 앞은 행(샘플)의 수, 뒤는 열(속성)의 수
행의 수는 batch_size에서 정하기 때문에 특별히 세지 않음
Param : 파라미터 수 = 가중치 + 바이어스
Dense_1: node = 12, input = 8 =>
Trainable params : 학습을 하면서 업데이트 된 총 파라미터 개수
| 피마 인디언 당뇨병 예측 모델의 구조 |
|---|
 |
Chap 12. 다중 분류 문제 해결하기
- 아이리스 데이터의 샘플, 속성, 클래스 구분
| 정보 1 | 정보 2 | 정보 3 | 정보 4 | 품종 | |
|---|---|---|---|---|---|
| 1번째 아이리스 | 5.1 | 3.5 | 1.4 | 0.2 | Iris-setosa |
| 2번째 아이리스 | 4.9 | 3.0 | 1.4 | 0.2 | Iris-setosa |
| 3번째 아이리스 | 4.7 | 3.2 | 1.3 | 0.3 | Iris-setosa |
| ... | ... | ... | ... | ... | ... |
| 150번째 아이리스 | 5.9 | 3.0 | 5.1 | 1.8 | Iris-virginica |
- 샘플 수 : 150개
- 속성 수 : 4개
- 정보 1 : 꽃받침 길이(sepal length, 단위 : cm)
- 정보 2 : 꽃받침 저비(sepal width, 단위 : cm)
- 정보 3 : 꽃잎 길이(petal length, 단위 : cm)
- 정보 4 : 꽃잎 너비(petal width, 단위 : cm)
- 클래스 : Iris-setosa, Iris-versicolor, Iris-virginica
- 다중분류 : 여러 개의 답(클래스) 중 하나를 고르는 분류 문제
Python
import pandas as pd
df = pd.read_csv('./data/iris3.csv')
df.head()
import seaborn as sns
import matplotlib.pyplot as plt
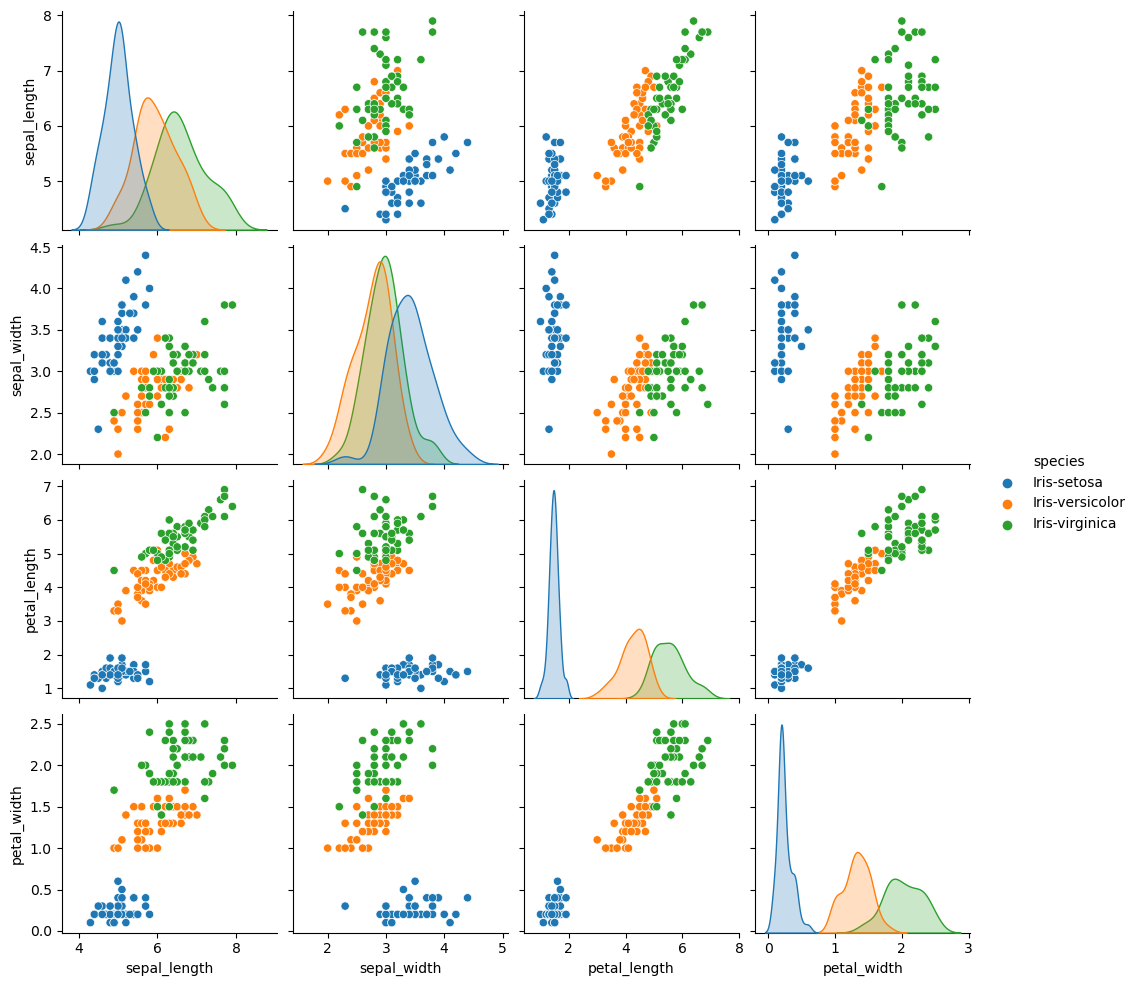
sns.pairplot(df, hue='species')
plt.show()
sns.pairplot(): 상관도 그래프,hue: 어떤 카테고리를 중심으로 그래프를 그릴 지 정하는 옵션
pairplot(hue = 'species')
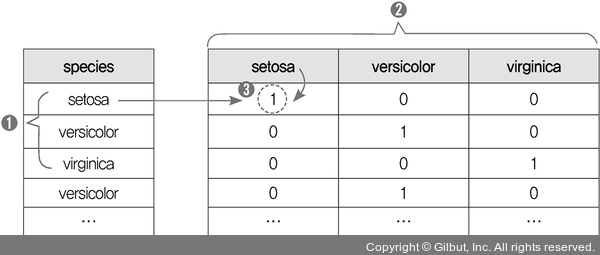
원-핫 인코딩
- 데이터에 글자(class)가 있으니 속성 부분과 클래스 부분으로 나누고
- 클래스 부분의 글자를 0과 1로 이루어진 형태로 바꿔줌 → 원-핫 인코딩(one-hot encoding)
- 각 클래스 별 확률을 따로 계산
Python
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
df = pd.read_csv('./data/iris3.csv')
X = df.iloc[:,0:4]
y = df.iloc[:,4]
y = pd.get_dummies(y)
model = Sequential()
model.add(Dense(12, input_dim=4, activation='relu'))
model.add(Dense(8, activation='relu'))
model.add(Dense(3, activation='softmax'))
model.summary()
model.compile(loss='categorical_crossentropy', optimizer='adam', metrics=['accuracy'])
history=model.fit(X, y, epochs=30, batch_size=5)다음과 같은 csv 표를 모두 숫자로 바꿔주기 위해 품종(class)(5번째 열)만 따로 때어냄
정보 1 정보 2 정보 3 정보 4 품종 1번째 아이리스 5.1 3.5 1.4 0.2 Iris-setosa 2번째 아이리스 4.9 3.0 1.4 0.2 Iris-setosa 3번째 아이리스 4.7 3.2 1.3 0.3 Iris-setosa ... ... ... ... ... ... 150번째 아이리스 5.9 3.0 5.1 1.8 Iris-virginica
.get_dummies()함수로 아래와 같이 원-핫 인코딩 처리함.(LUT과 비슷한 방식)
one-hot encoding 첫 번째 층의 input : 4개, node : 12개
두 번째 층의 node : 8개
마지막 층의 output : 3개, 활성화 함수 : softmax
손실 함수 : categorical_crossentropy
- softmax : 각 항목당 예측 확률을 0과 1 사이의 값으로 나타내주는데, 총합이 1이 된다.
- 이항 분류 :
binary_crossentropy, 다항 분류 :categorical_crossenttopy