
① Iris Data 를 통한 PCA 실습
② Wine Data를 통한 PCA 실습
③ plt, sns, plotly.express 비교
Iris Data - PCA 실습
Iris Data 불러오기
sklearn의Datasets에 저장된iris데이터를 불러온다.- 기존 진행시와 같이,
Pandas의DataFrame형태로 변경
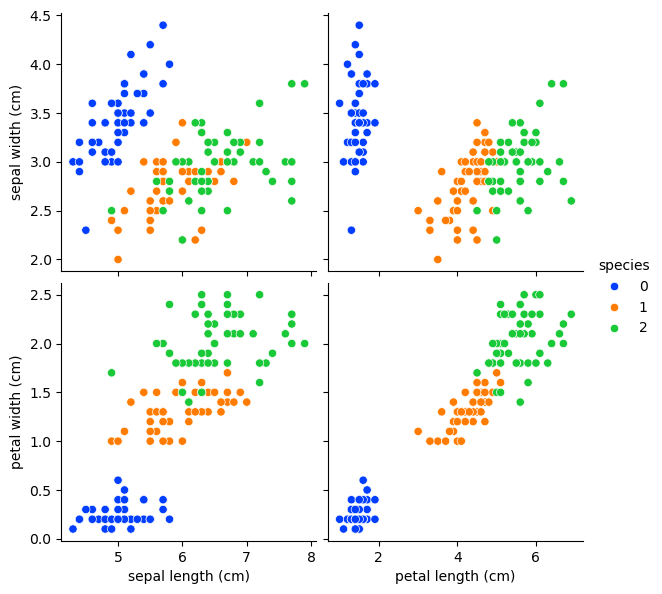
- 그래프로 그려서 각 특성(
feature)에 대한 라벨값(정답값)을 확인해보면 위와 같다
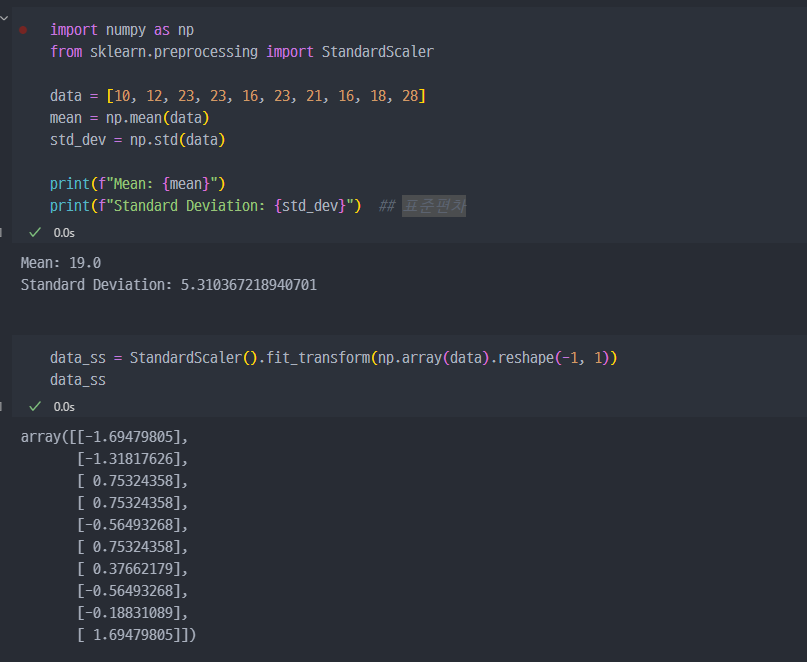
StandardScaler
Standard Scaler관련 내용은 이전 강의자료 참조. Standard_Scaler
각
feature들의 값의 범위가 다양하므로..petal width의 경우 0.2, 0.1, 등... 이루어져 있으며,
sepal length의 경우 5.1, 4.9 등 그 단위가 상이하다.
이를 일정하게 조정해주는것이
Scaler이다.
특히나
PCA분석의 경우분산에 많은 영향을 미치며, 스케일이 큰 값들의 경우 주성분으로 분류될 소지가 있다. 그렇기때문에StandardScaler를 적용해서 각feature들의 스케일조정을 진행해야 한다.
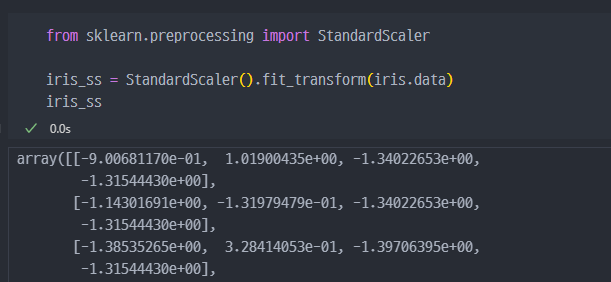
irisdata에StandardScaler적용 진행
Standard Scaler참고자료
PCA 진행
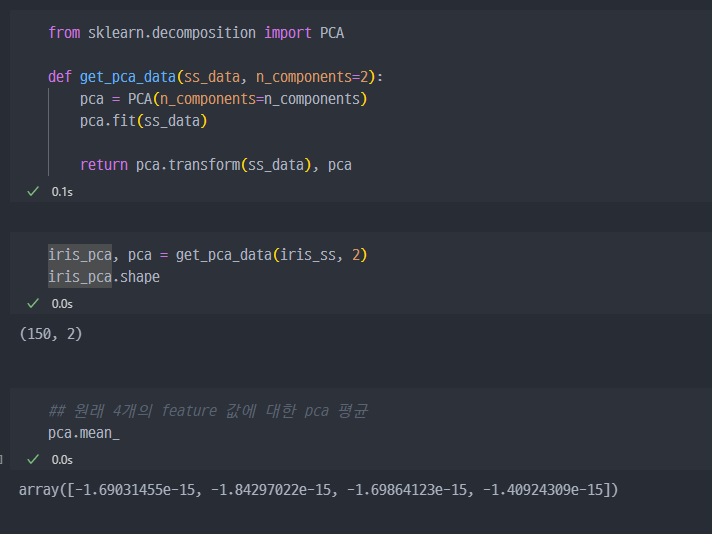
PCA진행
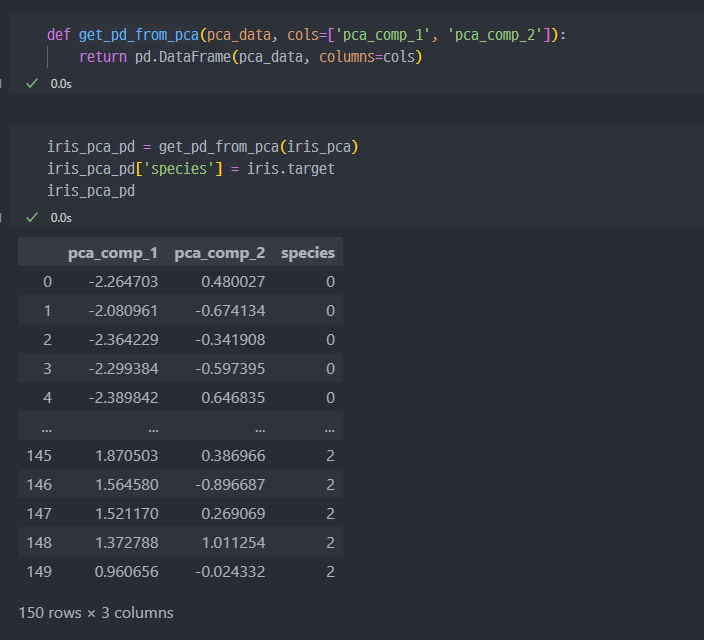
PCA결과값 및 결과를Pandas의 DataFrame으로 변경
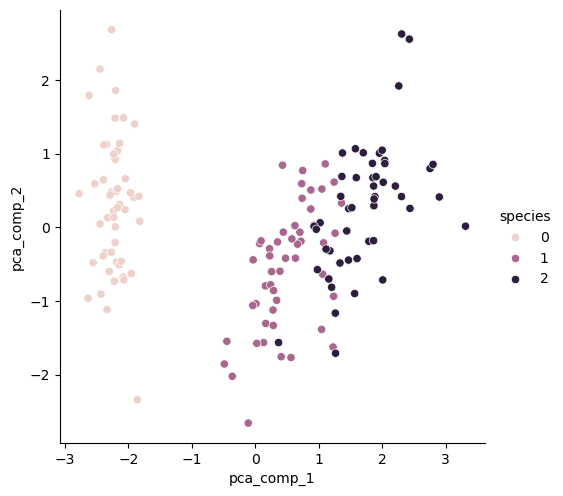
pairplot으로pca된 결과 표시
PCA로 RandomForest 진행
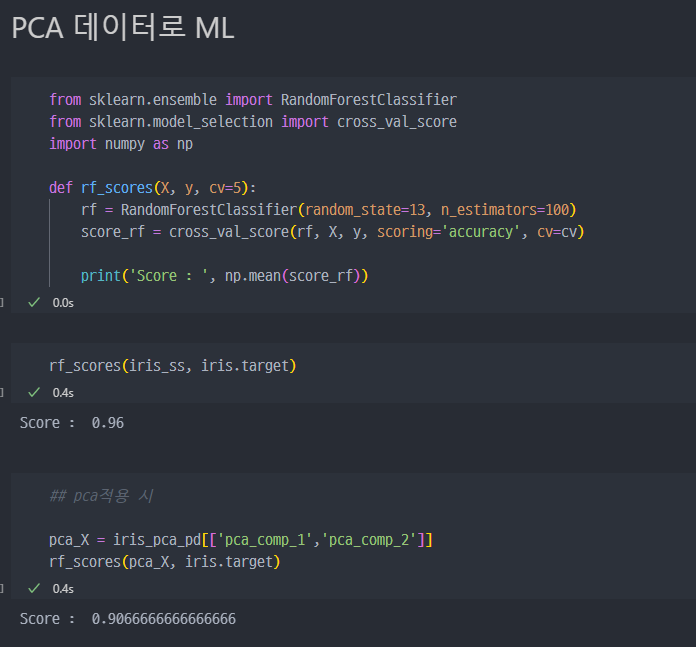
RandomForest를 이용하여val_score의Accuray확인
전체 데이터를 이용하는 원본(iris_ss)이 수치가 조금 더 좋은 것을 확인할 수 있음.
pca진행 시 아무래도 데이터의 축소가 이루어지고,component역시 2개만 선택되는 영향인 것 같음.
Wine Data - PCA 실습
데이터 불러오기
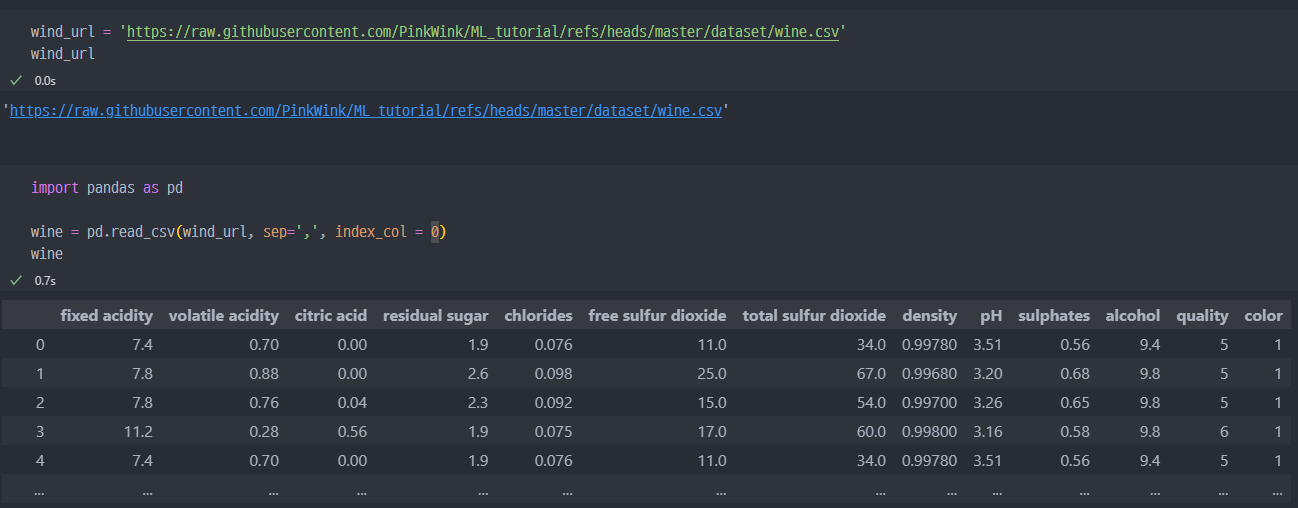
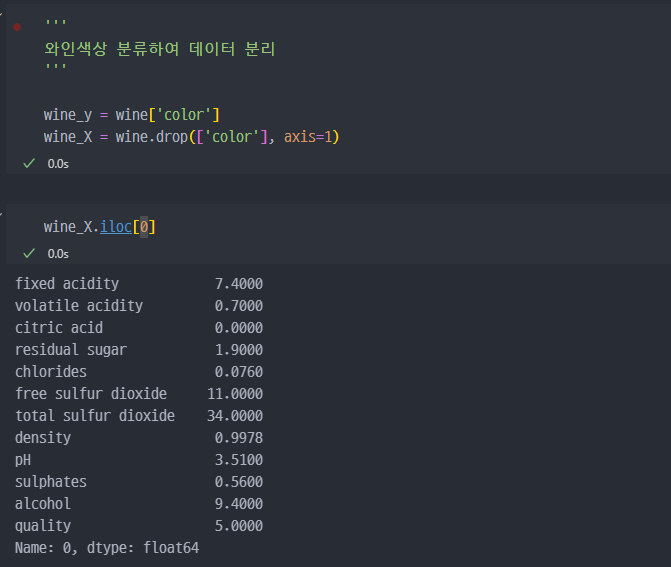
기존에 했던것처럼,
GitHub에서rawdata불러오기 진행.feature데이터와label데이터 분리
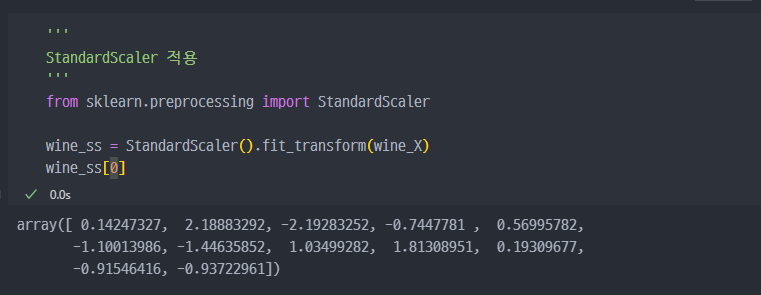
StandardScaler 적용
데이터값을 보면, 0.7 단위부터 34 까지 각 feature별로 다양하므로 PCA 적용을 위해 먼저 StandardScaler 적용
PCA 적용
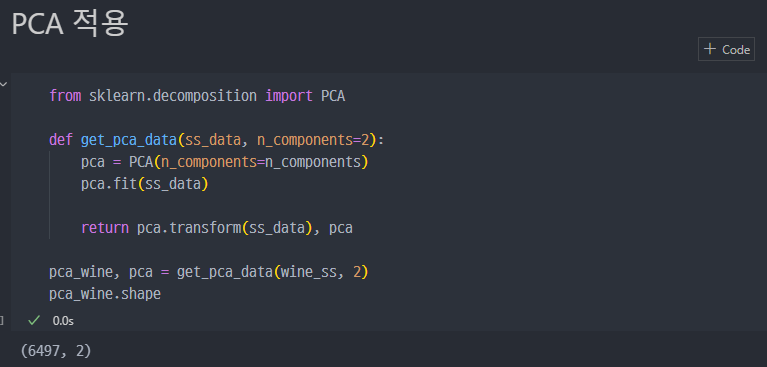
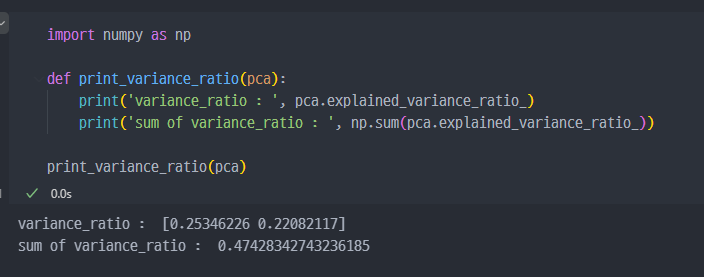
return 값으로 pca된 데이터 및 pca 객체를 받는다.
주성분을 2로 진행하였더니 50%가 되지 않는다..
pca한 데이터를pandas의 DataFrame 형태로 변경. (라벨값 포함)
PCA데이터 - pairplot
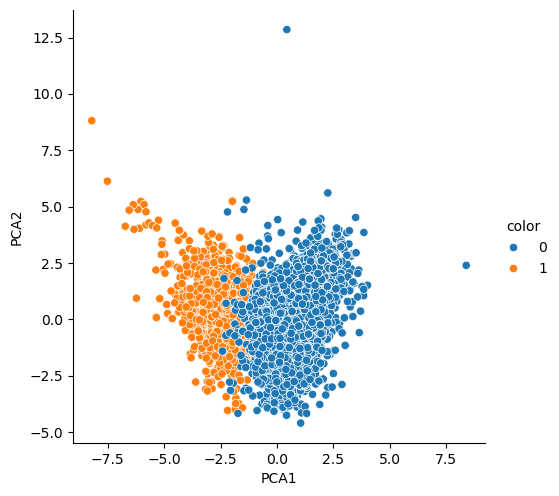
중앙에 조금 섞여있는 데이터들이 있지만, 대체로 잘 나누어진 듯?
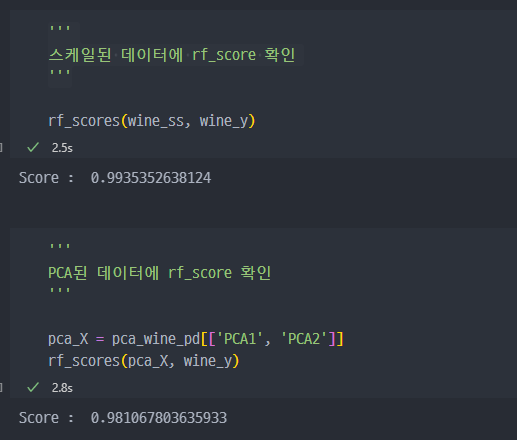
wine_ss와pca_X에 대한 rf_score 확인 => 큰 차이는 없는듯
n_component=3 로 분석진행
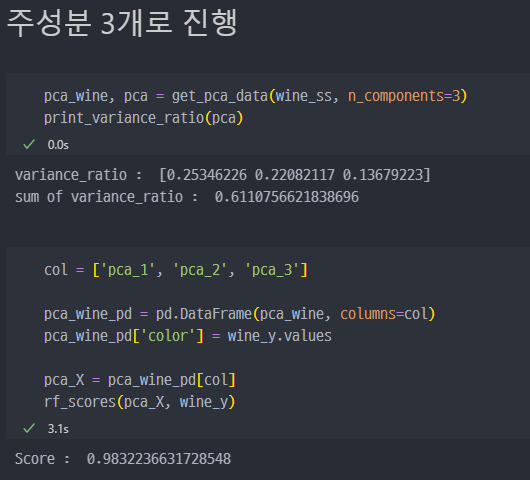
주성분 (
n_components=3)로 지정하여 분석진행.
주성분이 2개일 때와rf_score값은 큰 차이 없어보인다.variance_ratio는 조금 상승하였다.
그래프로 한번 바라보면...
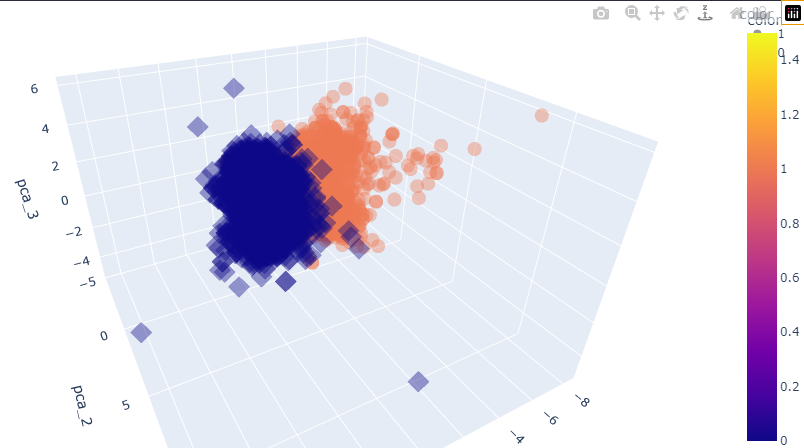
ploty.express VS plt VS sns
| 구 분 | Plotly.Express | Matplotlib.pyplot | Seaborn |
|---|---|---|---|
| InterActive 지원 | 강력 (줌, 팬, 툴팁 등) | 제한적 | 제한적 |
| 사용 난이도 | 쉬움(자동설정) | 중간~높음(설정 필요) | 중간 (기본설정 간편) |
| 커스터마이징 | 제한적 | 매우 강력 | matplotlib 기반 |
| 데이터 소스 | Pandas, JSON 지원(제한적) | 매우 강력 | matplotlib 기반 |
| 시각화 | 쏘쏘 | 논문용 | Good |